Deubiquitinase
Deubiquitinases (DUBs) are a large group of proteases that cleave ubiquitin from proteins and other molecules. Ubiquitin is attached to proteins in order to regulate the degradation of proteins via the proteasome and lysosome; coordinate thecellular localisation of proteins; activate and inactivate proteins; and modulate protein-protein interactions. DUBs can reverse these effects by cleaving the peptide or isopeptide bond between ubiquitin and its substrate protein. In humans there are nearly 100 DUB genes, which can be classified into two main classes: cysteine proteases and metalloproteases. The cysteine proteases comprise ubiquitin-specific proteases (USPs), ubiquitin C-terminal hydrolases (UCHs), Machado-Josephin domain proteases (MJDs) and ovarian tumour proteases (OTU). The metalloprotease group contains only the Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM) domain proteases. DUBs play several roles in the ubiquitin pathway. One of the best characterised functions of DUBs is the removal of monoubiqutin and polyubiquitin chainsfrom proteins.
Targets for Deubiquitinase
Products for Deubiquitinase
- Cat.No. Nom du produit Informations
-
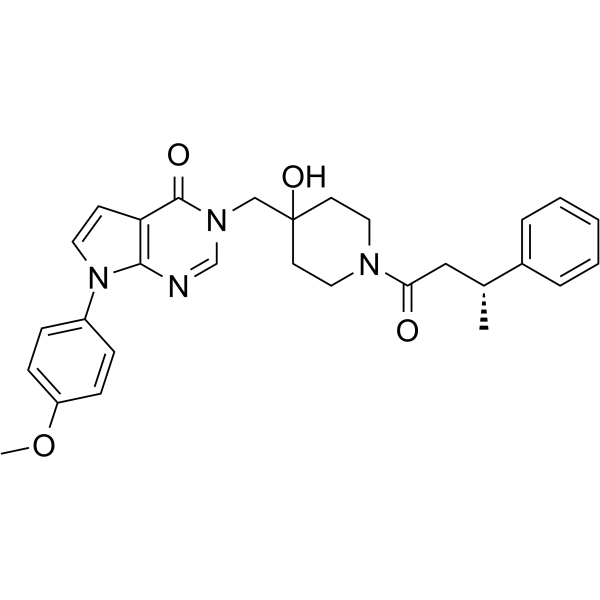
GC39625
6RK73
6RK73 est un inhibiteur covalent irréversible et spécifique de l'UCHL1 avec une IC50 de 0,23 μM. 6RK73 ne montre presque aucune inhibition de UCHL3 (IC50 = 236 μM). 6RK73 inhibe spécifiquement l'activité UCHL1 dans le cancer du sein.
-
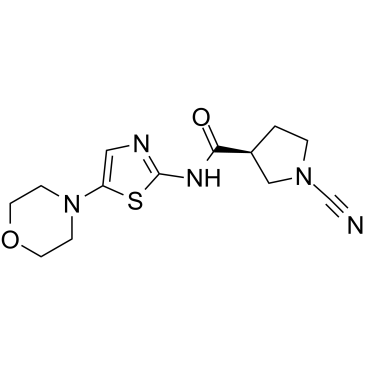
GC13035
Bay 11-7821
BAY 11-7082
Bay 11-7821(BAY 11-7082) est un inhibiteur de la phosphorylation de l'IκBα et du NF-κB, inhibant de manière sélective et irréversible la phosphorylation de l'IκB-α induite par le TNF-α (valeur IC50 d'environ 10 μM), et réduit l'expression de NF-κB ainsi que des molécules d'adhésion. Le Bay 11-7821 inhibe les protéases spécifiques de l'ubiquitine USP7 et USP21 avec des valeurs IC50 de 0,19 et 0,96 μM, respectivement.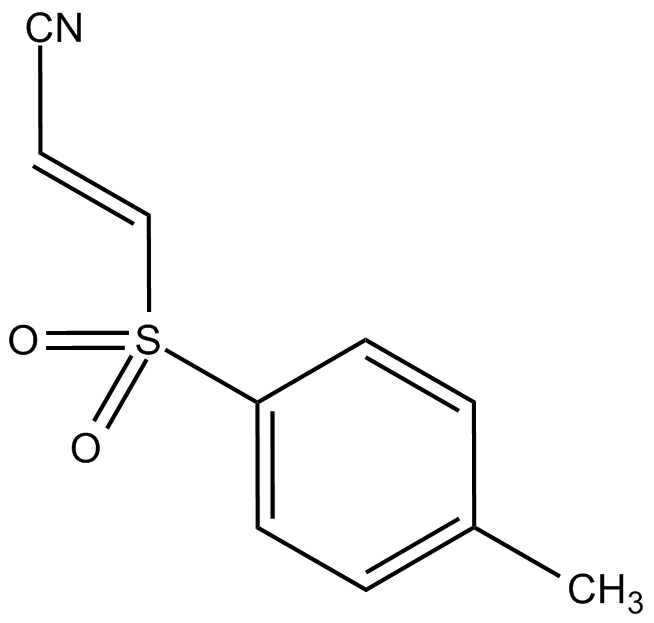
-
GC68740
BAY-728
BAY-728 peut être utilisé comme contrôle négatif de BAY-805. BAY-805 est un inhibiteur sélectif efficace de l'USP21.

-
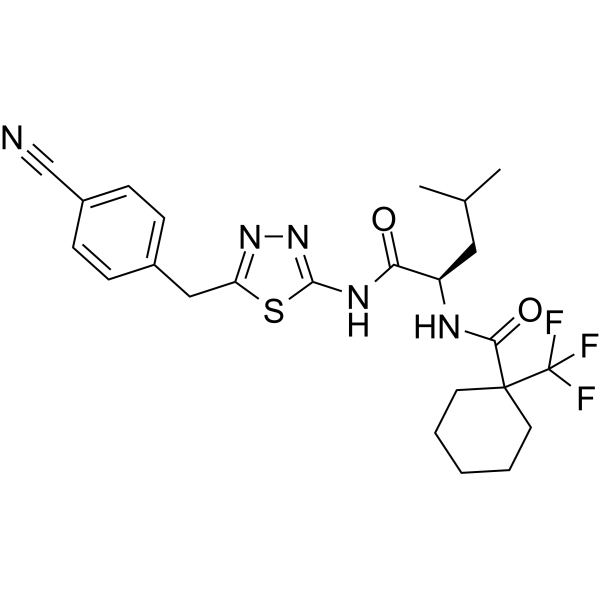
GC68742
BAY-805
BAY-805 est un inhibiteur sélectif de l'enzyme protéase spécifique à l'ubiquitine USP21. BAY-805 présente une haute sélectivité pour les cibles d'enzyme de déubiquitination (DUB), les kinases, les protéases et autres enzymes hors-cible courantes.
-
GC65259
BC-1471
BC-1471 est un inhibiteur de la déubiquitinase STAM-binding protein (STAMBP).

-
GC13892
C527
C527 est un inhibiteur enzymatique pan DUB, avec une puissance élevée pour le complexe USP1/UAF1 (IC50 = 0,88 μM).
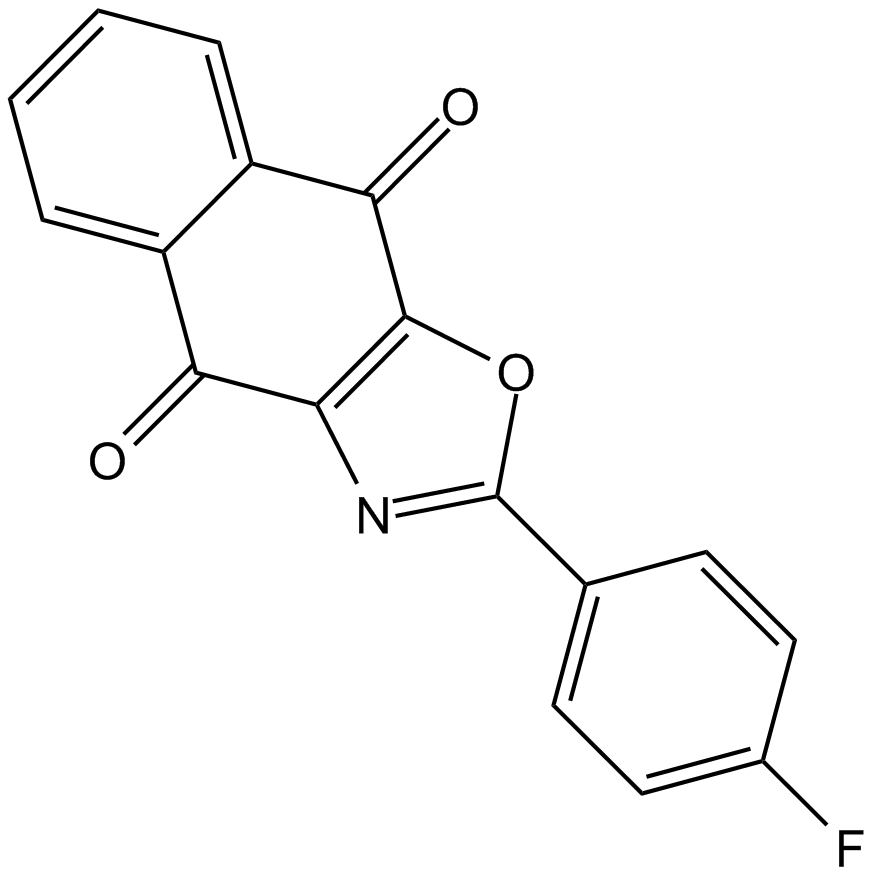
-
GC67886
DC-U4106
-
GC35906
DUBs-IN-1
DUBs-IN-1 est un inhibiteur actif des protéases spécifiques À l'ubiquitine (USP), avec une IC50 de 0,85 μM pour USP8.

-
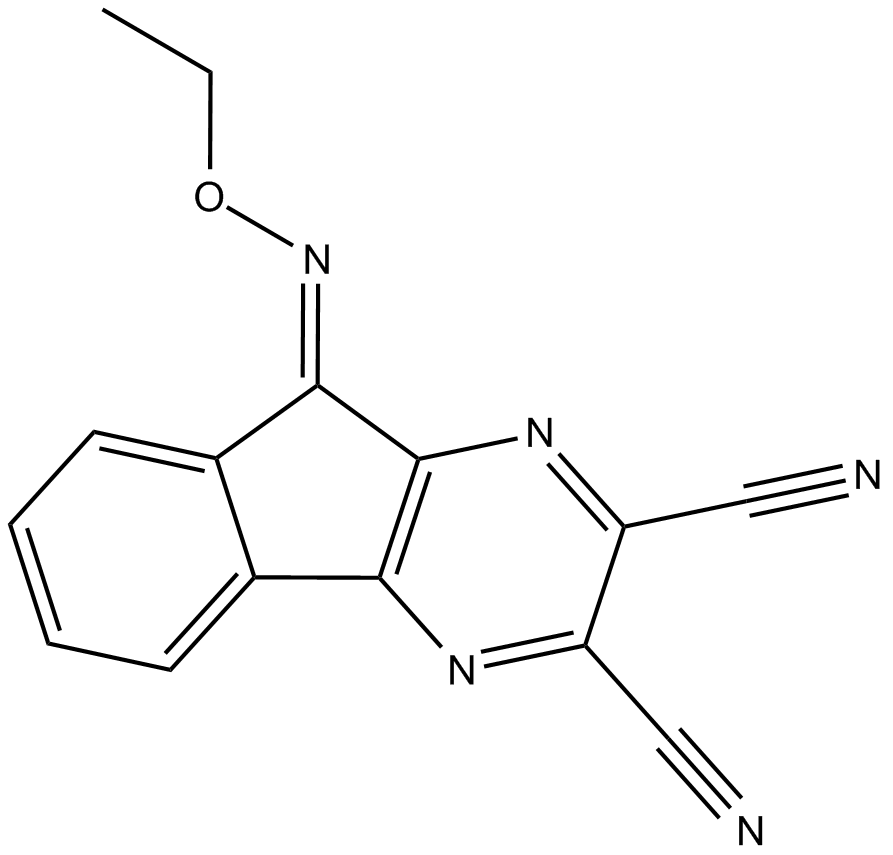
GC17125
DUBs-IN-2
DUBs-IN-2 est un puissant inhibiteur de la déubiquitinase avec une IC50 de 0,28 μM pour USP8.
-
GC10356
DUBs-IN-3
DUBs-IN-3 est un puissant inhibiteur de l'enzyme déubiquitinase (USP) extrait du composé de référence 22c avec une IC50 de 0,56 μM pour USP8.

-
GC34083
EOAI3402143
EOAI3402143 est un inhibiteur de la déubiquitinase (DUB), qui inhibe de manière dose-dépendante Usp9x/Usp24 et Usp5.

-
GC62979
FT206
Le FT206 est un inhibiteur des carboxamides comme la protrase spécifique de l'ubiquitine extrait du brevet WO 2020033707 A1, exemple 11-1.

-
GC67915
FT3967385
FT385

-
GC32786
FT671
FT671 est un inhibiteur puissant, non covalent et sélectif de l'USP7 avec une IC50 de 52 nM et se lie au domaine catalytique de l'USP7 avec un Kd de 65 nM.

-
GC69134
FT709
FT709 est un inhibiteur sélectif efficace de l'USP9X, avec une valeur IC50 de 82 nM. L'USP9X est impliqué dans les fonctions du centrosome, l'arrangement des chromosomes pendant la mitose, la dégradation du récepteur EGF, la sensibilité chimique et le rythme circadien.

-
GC32917
FT827
FT827 est un inhibiteur sélectif et covalent de la protéase 7 spécifique de l'ubiquitine (USP7) (Ki=4,2 ?M). FT827 se lie au domaine catalytique USP7 (USP7CD ; résidus 208-560) avec une valeur apparente de Kd de 7,8 ?M.

-
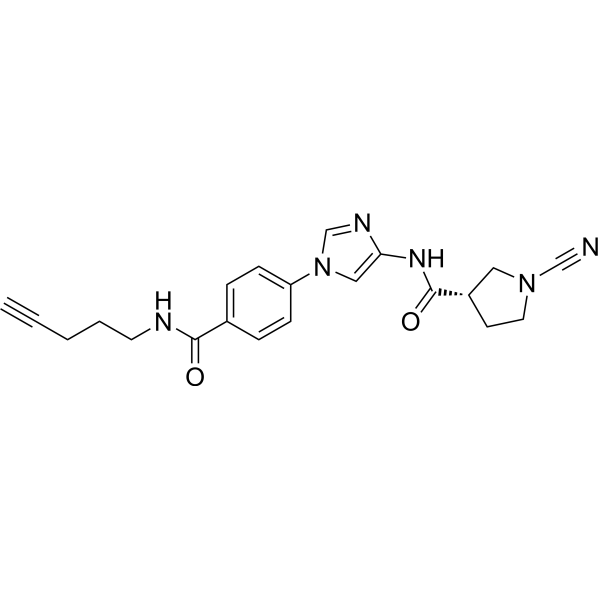
GC73465
GK13S
GK13S est un Ligand et inhibiteur de l'ubiquitinase uchl1.
-
GC71305
GK16S
GK16S est une sonde chimiogénomique UCHL1.
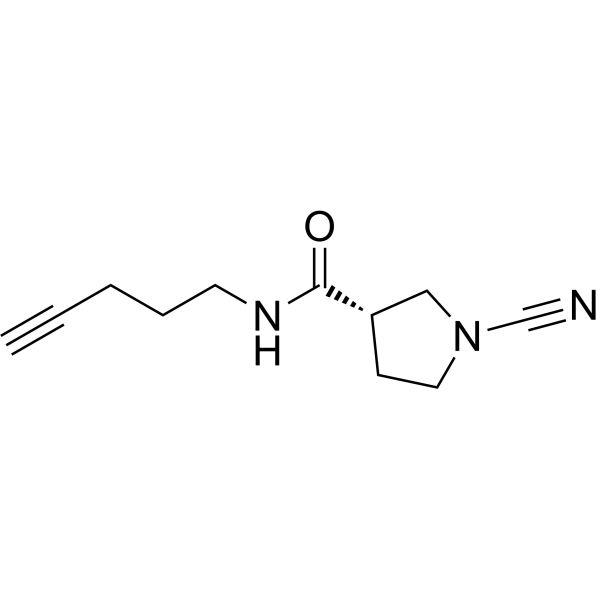
-
GC32856
GNE-6640
GNE-6640 est un inhibiteur sélectif et non covalent de la peptidase spécifique À l'ubiquitine 7 (USP7), avec des valeurs IC50 de 0,75 μM, 0,43 μM, 20,3 μM et 0,23 μM pour USP7 pleine longueur, domaine catalytique USP7, pleine longueur USP43 et Ub- MDM2, respectivement.

-
GC32727
GNE-6776
GNE-6776 est un inhibiteur sélectif et biodisponible de l'USP7 par voie orale.

-
GC39172
GRL0617
Un inhibiteur de la PLpro du SARS-CoV et du SARS-CoV-2.

-
GC34354
GSK2643943A
GSK2643943A est un inhibiteur de l'enzyme de déubiquitination (DUB) ciblant l'USP20. GSK2643943A a une affinité avec une IC50 de 160 nM pour USP20/Ub-Rho. GSK2643943A a une efficacité antitumorale et peut être utilisé pour la recherche sur le carcinome épidermoÏde oral (OSCC).

-
GC36211
HBX 19818
HBX 19818 est un inhibiteur spécifique de la protéase 7 spécifique À l'ubiquitine (USP7), avec une IC50 de 28,1 μM.

-
GC69249
I-138
I-138 est un composé oral efficace structurellement lié à ML323. I-138 et ML323 sont des inhibiteurs réversibles efficaces de l'interaction USP1-UAF1. I-138 montre une liaison synergique avec l'ubiquitine et une liaison mutuellement exclusive avec ML323. I-138 induit la monoubiquitination de FANCD2 et PCNA dans les cellules MDA-MB-436, augmentant la monoubiquitination de PCNA et FANCD2 dans les cellules HAP-1 USP1 WT. I-138 élimine l'autocatalyse d'USP1 dans les cellules.
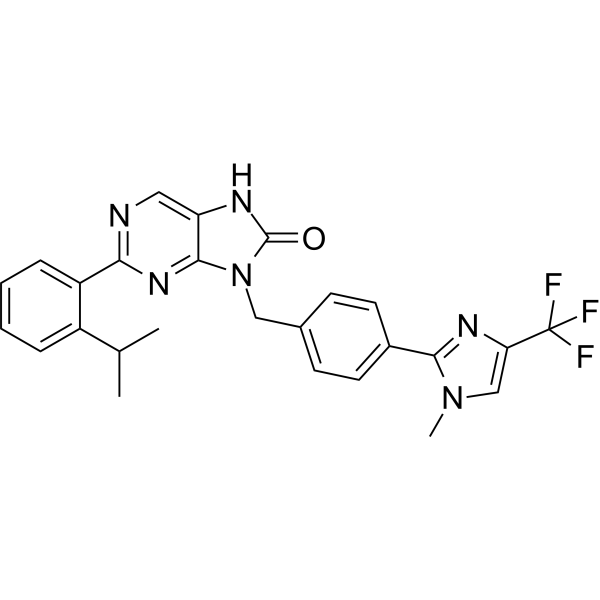
-
GC14797
IU1
Usp14 inhibitor
IU1 est un inhibiteur sélectif réversible et cytoper cyto - perméable du protéasome, ce que IC50 est 4,7µM pour USP14.
-
GC63027
IU1-248
IU1-248, un dérivé de IU1, est un inhibiteur puissant et sélectif de l'USP14 avec une IC50 de 0,83μM.

-
GC64956
IU1-47
IU1-47 est un inhibiteur puissant et spécifique de l'USP14 avec une IC50 de 0,6 μM.

-
GC62460
LCAHA
LCA hydroxyamide
LCAHA (LCA hydroxyamide) est un inhibiteur de la déubiquitinase USP2a avec des IC50 de 9,7 μM et 3,7 μM dans les tests Ub-AMC et Di-Ub, respectivement. LCAHA déstabilise la cycline D1 et induit l'arrêt G0/G1 en inhibant la déubiquitinase USP2a.
-
GC10510
LDN 57444
Ubiquitin C-terminal Hydrolase L1 Inhibitor, UCHL1 Inhibitor
An inhibitor of UCH-L1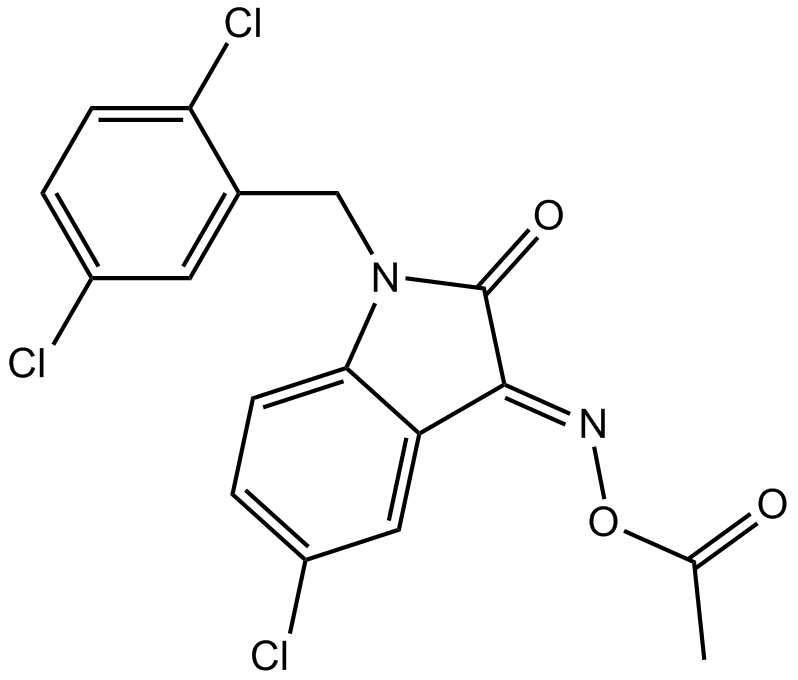
-
GC60981
LDN-91946
Le LDN-91946 est un inhibiteur puissant, sélectif et non compétitif de l'hydrolase C-terminale de l'ubiquitine-L1 (UCH-L1) avec un Ki app de 2,8 μM.

-
GC34670
MF-094
Le MF-094 est un inhibiteur puissant et sélectif de l'USP30 avec une IC50 de 120 nM. Le MF-094 augmente l'ubiquitination des protéines et accélère la mitophagie.
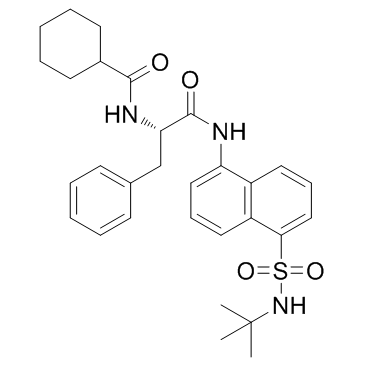
-
GC17635
ML-323
Le ML-323 est un puissant inhibiteur réversible de l'USP1-UAF1 avec une IC50 de 76 nM dans un test Ub-Rho. Les constantes d'inhibition mesurées du ML-323 pour l'enzyme libre (Ki) sont de 68 nM.
-
GC32720
ML364
ML364 est un inhibiteur sélectif de la peptidase 2 spécifique de l'ubiquitine (USP2) (IC50 = 1,1 μM) avec une activité anti-proliférative, qui se lie directement À l'USP2 (Kd = 5,2 μM), induit une augmentation de la dégradation de la cycline D1 cellulaire et provoque un arrêt du cycle cellulaire. ML364 augmente les niveaux de ROS mitochondriales et diminue le contenu intracellulaire de l'ATP.

-
GC12475
NSC 632839 hydrochloride
F6, Ubiquitin Isopeptidase Inhibitor II
A deubiquitylase and deSUMOylase inhibitor
-
GC15503
NSC 687852 (b-AP15)
b-AP15
An inhibitor of the deubiquitinases USP14 and UCHL5
-
GC69637
OTUB1/USP8-IN-1
OTUB1/USP8-IN-1 est un inhibiteur double efficace d'OTUB1/USP8, avec des valeurs IC50 d'inhibition respectives de 0,17 et 0,28 nM pour OTUB1 et USP8. OTUB1/USP8-IN-1 peut être utilisé dans la recherche sur le cancer.

-
GC73354
OTUB1/USP8-IN-1 TFA
OTUB1/USP8-IN-1 TFA est la forme de sel TFA de OTUB1/USP8-IN-1.

-
GC71471
OTUB2-IN-1
OTUB2-IN-1, a specific inhibitor of OTUB2 (KD: ~12 μM), reduces PD-L1 protein expression in tumor cells and inhibits tumor growth by promoting robust intra-tumor infiltration of cytotoxic T lymphocytes (CTL) .
-
GC10379
P 22077

-
GC12067
P005091
P005091,P5091
Le P005091 est un inhibiteur puissant et sélectif de la protéinase spécifique de l'ubiquitine 7 (USP7) avec une valeur EC50 de 4,2 μM.
-
GC13208
PR-619
Le PR-619 est un inhibiteur à large spectre des enzymes déubiquitinantes (DUB). Le PR-619 a démontré une activité inhibitrice significative des DUB (5-20 µM) et une activité inhibitrice de la croissance avec une IC50 de 2 µM dans les cellules HEK 293T.
-
GC60317
RA-9
RA-9 est un inhibiteur puissant et sélectif des enzymes de désubiquitination associées au protéasome (DUB) avec un profil de toxicité favorable et une activité anticancéreuse. RA-9 bloque la dégradation des protéines dépendante de l'ubiquitine sans affecter l'activité protéolytique du protéasome 20S. RA-9 induit sélectivement l'apparition de l'apoptose dans les lignées cellulaires du cancer de l'ovaire et les cultures primaires dérivées de donneurs. RA-9 induit des réponses de stress du réticulum endoplasmique (ER) dans les cellules cancéreuses de l'ovaire.

-
GC17698
SJB2-043
An inhibitor of the USP1-UAF1 complex and SARS-CoV-2 PLpro

-
GC13401
SJB3-019A
SJB3-019A est un puissant et nouvel inhibiteur de l'USP1, 5 fois plus puissant que SJB2-043 pour favoriser la dégradation et la cytotoxicité de l'ID1 dans les cellules K562 avec une IC50 de 0,0781 μM.

-
GC67712
STAMBP-IN-1

-
GC62509
STD1T
STD1T est un inhibiteur de la déubiquitinase USP2a avec une IC50 de 3,3 μM dans le test Ub-AMC.

-
GC13013
TCID
UCH-L3 Inhibitor
Le TCID (4,5,6,7-tétrachloroindan-1,3-dione) est un inhibiteur puissant et sélectif de l'hydrolase C-terminale de l'ubiquitine neuronale (UCH-L3) avec une IC50 de 0,6 μM.
-
GC14366
Thioguanine
NSC 752, NSC 76504, 6TG
A thiopurine analog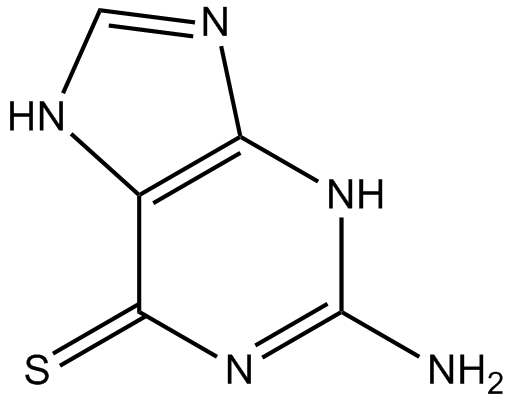
-
GC73166
U7D-1
U7D-1 est un dégradeur PROTAC puissant et sélectif de première classe USP7 (ubiquitin-specific protease 7), avec un DC50 de 33 nM dans les cellules RS4;11.
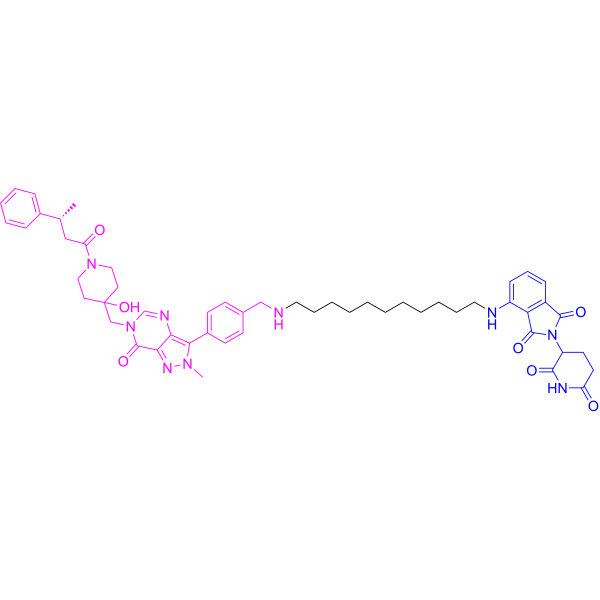
-
GC73948
UCHL1-IN-1
UCHL1-IN-1 (composé 46) est un inhibiteur de l’ubiquitine c-drolase terminale L1 (UCHL1), avec une ci50 de 5,1 μM.
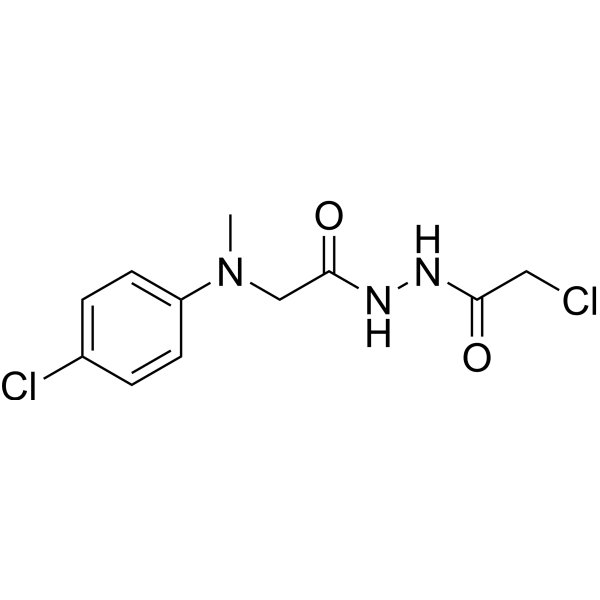
-
GC70098
USP1-IN-2
USP1-IN-2 (composé I-193) est un inhibiteur efficace de l'USP1, avec une IC50 inférieure à 50 nM.

-
GC73156
USP1-IN-3
USP1-IN-3 est un inhibiteur USPI sélectif.
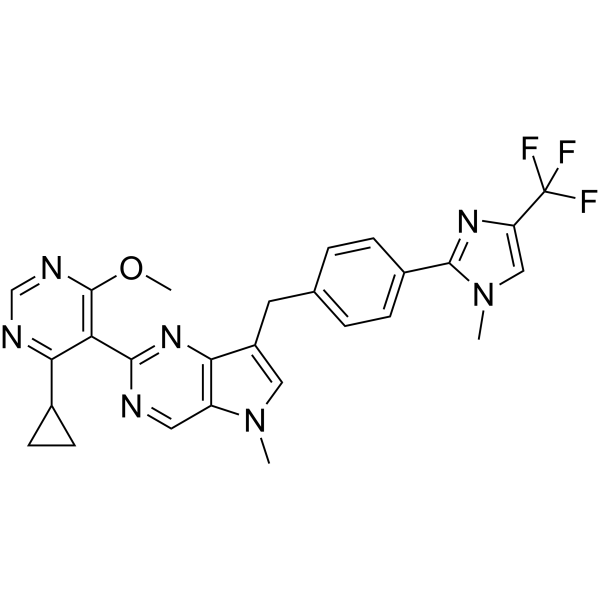
-
GC67963
USP15-IN-1

-
GC38043
USP25/28 inhibitor AZ1
AZ1
USP25/28 inhibitor AZ1 (AZ1) est un inhibiteur double, non compétitif, sélectif et actif par voie orale de la protéase spécifique de l'ubiquitine (USP) 25/28 avec des IC50 de 0,7 μM et 0,6 μM, respectivement.
-
GC73221
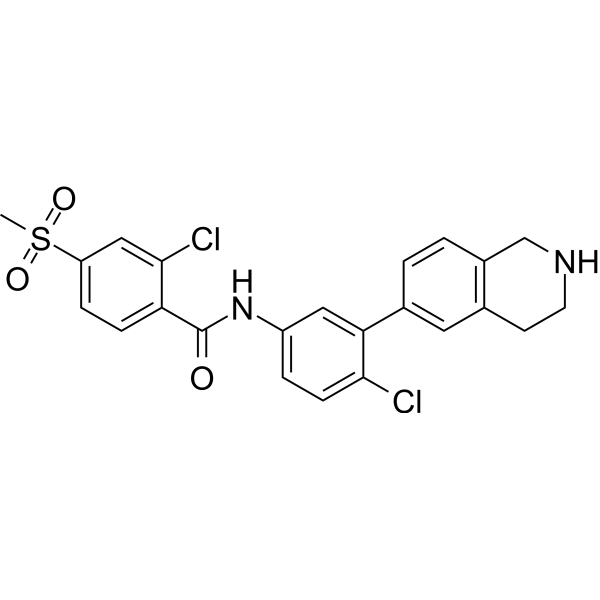
USP28-IN-3
USP28-IN-3 est un inhibiteur USP28 (IC50= 0,1 μM) avec une sélectivité élevée sur USP2, USP7, USP8, USP9x, UCHL3 et UCHL5.
-
GC73222
USP28-IN-4
USP28-IN-4 est un inhibiteur USP28 (IC50= 0,04 μM) avec une sélectivité élevée sur USP2, USP7, USP8, USP9x, UCHL3 et UCHL5.

-
GC70099
USP30 inhibitor 11
L'inhibiteur USP30 11 est un inhibiteur sélectif et efficace de l'USP30, avec une valeur IC50 de 0,01 µμ. L'inhibiteur USP30 11 est le composé numéro 83 issu du document de brevet WO2017009650A1, utilisé pour étudier les cancers et les maladies liées aux dysfonctionnements mitochondriaux.

-
GC62410
USP30 inhibitor 18
L'inhibiteur USP30 18 est un inhibiteur sélectif de l'USP30 avec une IC50 de 0,02 μM. L'inhibiteur USP30 18 augmente l'ubiquitination des protéines et accélère la mitophagie.

-
GC66438
USP5-IN-1
USP5-IN-1 (composé 64), un puissant inhibiteur de l'enzyme déubiquitinase USP5, se lie au domaine ZnF-UBD de l'USP5 avec une KD de 2,8 μM. USP5-IN-1 est sélectif par rapport à neuf protéines contenant des domaines ZnF-UBD structurellement similaires. USP5-IN-1 inhibe la clivage catalytique de l'USP5 d'un substrat di-ubiquitine.

-
GC37875
USP7-IN-1
USP7-IN-1 est un inhibiteur sélectif et réversible de la protéase 7 spécifique À l'ubiquitine (USP7), avec une IC50 de 77 μM, et peut être utilisé pour la recherche sur le cancer.

-
GC73177
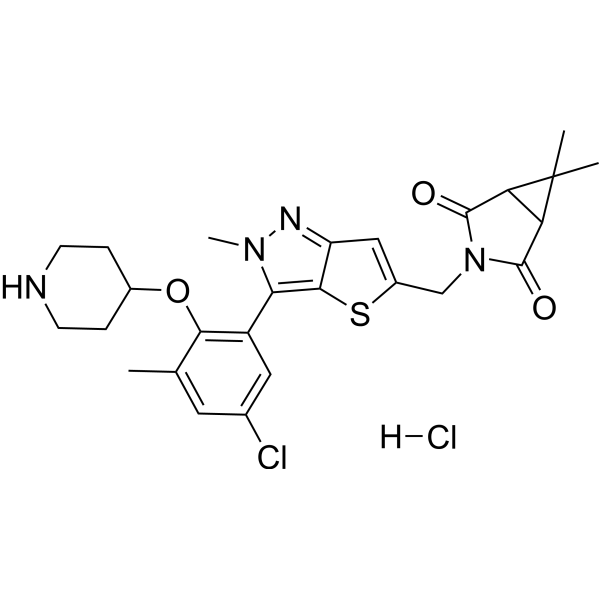
USP7-IN-10 hydrochloride
USP7-IN-10 hydrochloride (composé 1) est un puissant inhibiteur de la protéase 7 spécifique de l'Ubiquitine (usp7) avec une CI50 de 13,39 nm.
-
GC70100
USP7-IN-11
USP7-IN-11 est un inhibiteur efficace de la déubiquitinase spécifique à l'ubiquitine 7 (USP7), avec une IC50 de 0,37 nM. USP7-IN-11 a des effets anticancéreux (WO2022048498A1 ; exemple d'exécution 187).

-
GC73882
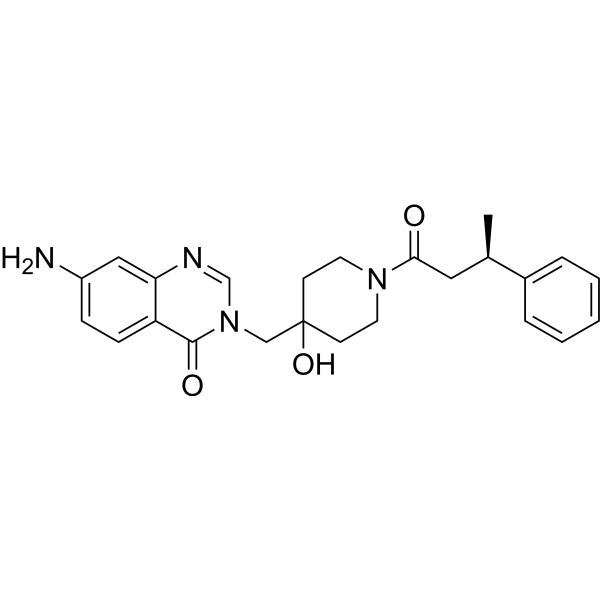
USP7-IN-13
USP7-IN-13 (composé 101) est un inhibiteur USP7 d’une valeur ci50 de 0,2-1 μM, qui peut être utilisé pour l’étude du myélome multiple.
-
GC65275
USP7-IN-3
L'USP7-IN-3 (composé 5) est un inhibiteur allostérique puissant et sélectif de la protéase 7 (USP7) spécifique de l'ubiquitine.

-
GC70101
USP7-IN-7
USP7-IN-7 (composé 124) est un inhibiteur de l'USP7 avec une valeur IC50 inférieure à 10 nM. USP7-IN-7 présente une toxicité cellulaire à faible nanomolarité pour les lignées cellulaires de cancer p53 muté, les tumeurs hématologiques p53 sauvages et les neuroblastomes. USP7-IN-7 peut être utilisé dans la recherche sur le cancer.

-
GC62377
USP7-IN-8
USP7-IN-8 (exemple 81) est un inhibiteur sélectif de la protéase 7 spécifique de l'ubiquitine (USP7) avec une CI50 de 1,4 μM dans un test Ub-Rho110. USP7-IN-8 ne montre aucune activité contre USP47 et USP5. USP7-IN-8 a des effets anticancéreux.

-
GC70102
USP7-IN-9
USP7-IN-9 is an efficient inhibitor of USP7 with an IC50 of 40.8 nM. It can induce apoptosis in RS4;11 cells and block the cell cycle at G0/G1 and S phase. USP7-IN-9 reduces the levels of cancer proteins MDM2 and DNMT1, while increasing the levels of tumor suppressor proteins p53 and p21.

-
GC18160
USP7/USP47 inhibitor
L'inhibiteur USP7/USP47 est un inhibiteur sélectif de la protéase 7/47 spécifique de l'ubiquitine (USP7/USP47), avec des CE50 de 0,42 μM et 1,0 μM, respectivement.

-
GC64537
USP8-IN-1
L'USP8-IN-1 est un inhibiteur de l'USP8 avec une IC50 de 1,9 μM. USP8-IN-1 inhibe la croissance des cellules H1975 avec un GI50 de 82,04 μM (CN111138358A ; U10).

-
GC73283
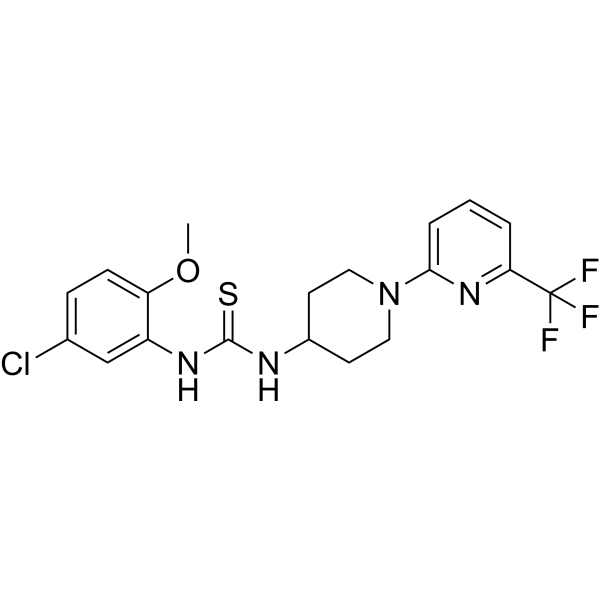
USP8-IN-2
USP8-IN-2 (Compd u52) est un inhibiteur de l'ubiquitinase usp8 avec une IC50 de 6,0 μm.
-
GC70103
USP8-IN-3
USP8-IN-3 (Composé U51) est un inhibiteur de l'enzyme déubiquitinase USP8, avec une valeur IC50 de 4,0 μM. USP8-IN-3 inhibe également la prolifération des cellules GH3 et H1957, avec des valeurs GI50 respectives de 37,03 μM et 6,01 μM.

-
GC19378
VLX1570
VLX1570 est un inhibiteur compétitif des deubiquitinases du protéasome (DUB) avec une IC50 d'environ 10 μM.

-
GC10970
WP1130
WP 1130; WP-1130
WP1130 (WP1130) est un inhibiteur de la déubiquitinase (DUB) perméable aux cellules, inhibant directement l'activité DUB de USP9x, USP5, USP14 et UCH37. Il a été démontré que WP1130 régule À la baisse les protéines anti-apoptotiques Bcr-Abl et JAK2.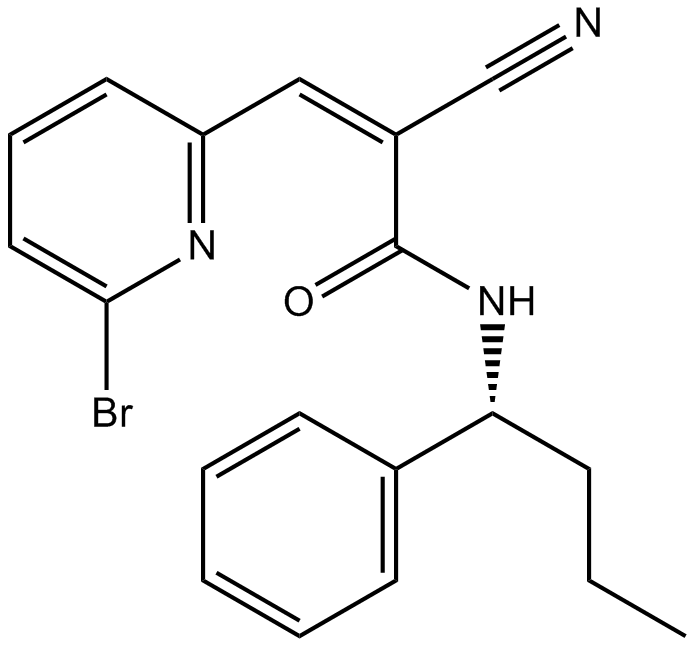
-
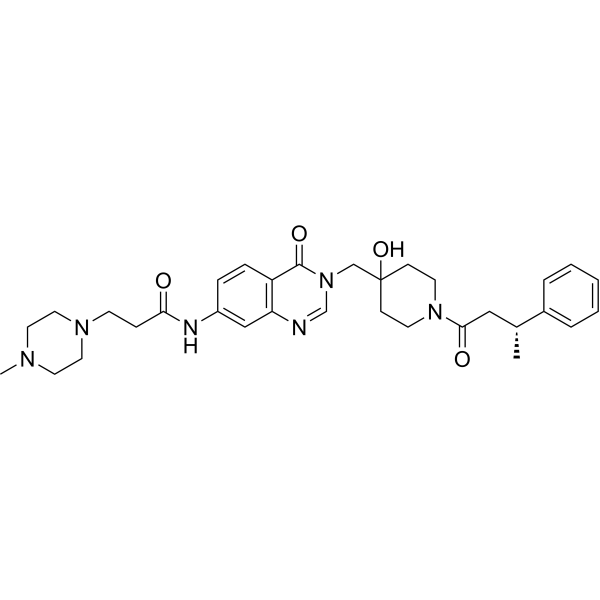
GC72934
XL 188
XL 188 est un inhibiteur puissant et sélectif de l'usp7 avec des valeurs IC50 de 90 nm et 193 nm pour l'enzyme usp7 pleine longueur et l'enzyme à domaine catalytique, respectivement.
-
GC62517
XL177A
XL177A est un inhibiteur USP7 irréversible très puissant et sélectif avec une IC50 de 0,34nM. XL177A provoque la destruction des cellules cancéreuses par un mécanisme dépendant de p53.

-
GC73774
YCH2823
YCH2823 est un inhibiteur d’usp7 (ci50 = 49,6 nM; Kd = 0,117 μM).