PARP
Poly (ADP-ribose) polymerases (PARPs) is a large family of proteins with a conserved catalytic domain that catalyze an immediate DNA-damage-dependent post-translational modification of histones and other nuclear proteins leading to the survival of injured proliferating cells. So far, a total number of 18 human PARP proteins encoded by different genes have been identified, including PARP-1 to PARP-4, PARP-5a, PARP-5b, PARP-5c and PARP-6 to PARP-16. The general structural of PARP proteins has been revealed through the extensive study of the founding family member PARP-1, which is characterized by the presence of four functional domains, including a DNA-binding domain, a caspase-cleaved domain, an automodification domain and a catalytic domain.
Products for PARP
- Cat.No. Nom du produit Informations
-
GC11761
4-amino-1,8-Naphthalimide
4-Aminonaphthalimide,4-ANI
Le 4-amino-1,8-naphtalimide est un puissant inhibiteur de PARP et potentialise la cytotoxicité des rayonnements γ-dans les cellules cancéreuses.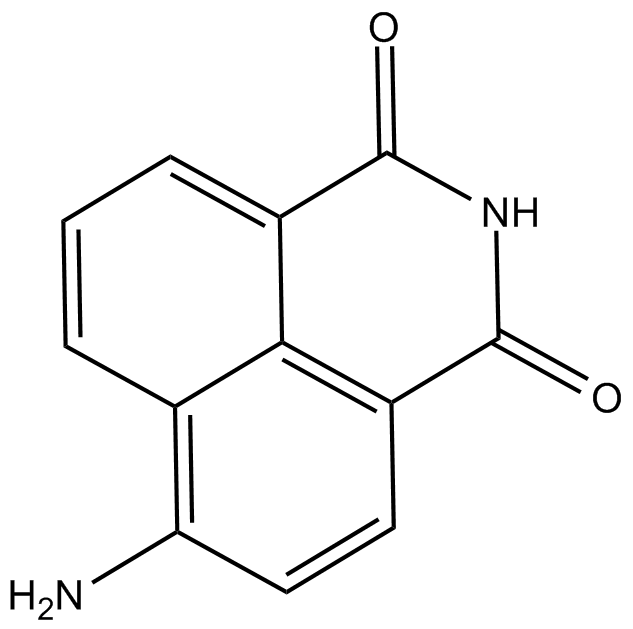
-
GC35150
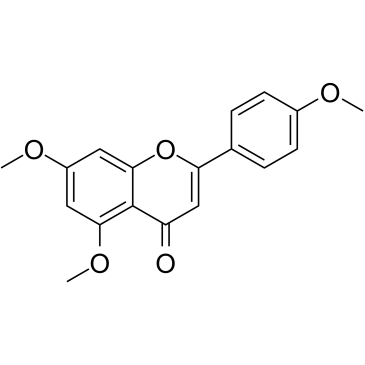
5,7,4'-Trimethoxyflavone
La 5,7,4'-triméthoxyflavone est isolée de Kaempferia parviflora (KP) qui est une célèbre plante médicinale de Thaïlande. La 5,7,4'-triméthoxyflavone induit l'apoptose, comme en témoignent les augmentations de la phase sous-G1, la fragmentation de l'ADN, la coloration annexine-V/PI, le rapport Bax/Bcl-xL, l'activation protéolytique de la caspase-3 et la dégradation de la poly (ADP-ribose) polymérase (PARP). La 5,7,4'-triméthoxyflavone est significativement efficace pour inhiber la prolifération des cellules cancéreuses gastriques humaines SNU-16 d'une manière dépendante de la concentration.
-
GC68161
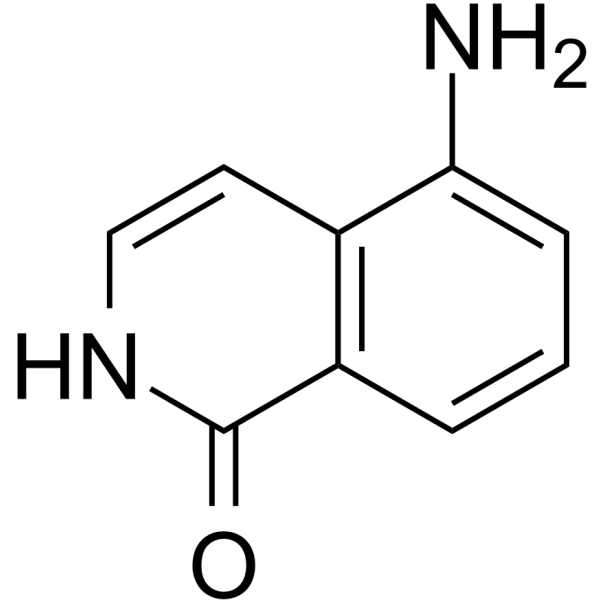
5-AIQ
5-Aminoisoquinolin-1-one
-
GC73642
ALK-IN-26
ALK-IN-26 est un inhibiteur d’alk d’une valeur ci50 de 7,0 μM pour l’alk tyrosine kinase.

-
GC70420
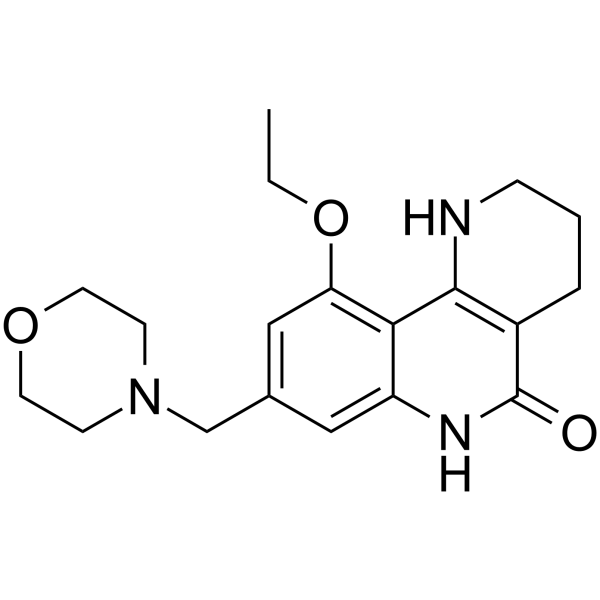
Amelparib
Amelparib est un inhibiteur puissant, actif par voie orale et soluble dans l'eau de PARP - 1.
-
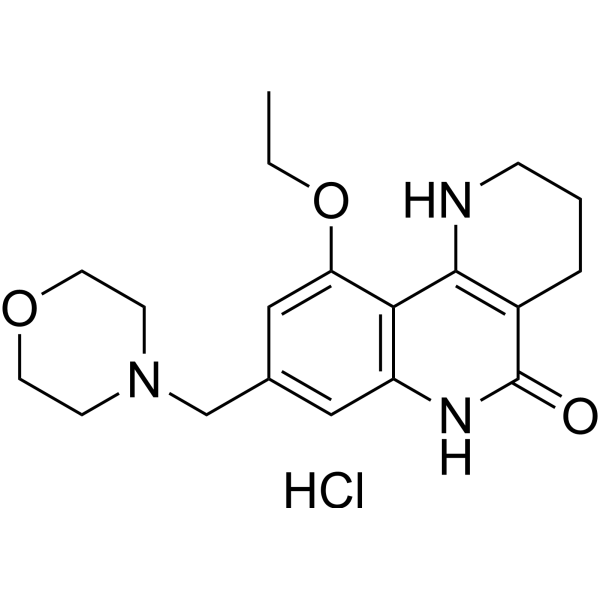
GC70421
Amelparib hydrochloride
Amelparib hydrochloride est un inhibiteur puissant, actif par voie orale et soluble dans l'eau de PARP - 1.
-
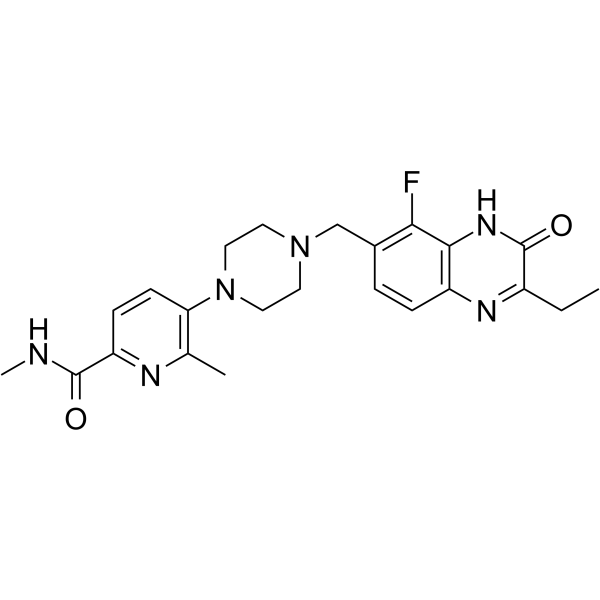
GC65899
AZ3391
AZ3391 est un puissant inhibiteur de PARP. AZ3391 est un dérivé de la quinoxaline. La famille d'enzymes PARP joue un rÔle important dans un certain nombre de processus cellulaires, tels que la réplication, la recombinaison, le remodelage de la chromatine et la réparation des dommages À l'ADN. AZ3391 a le potentiel pour la recherche de maladies et d'affections survenant dans les tissus du système nerveux central, tels que le cerveau et la moelle épinière (extrait du brevet WO2021260092A1, composé 23).
-
GC16725
AZ6102
AZ6102 est un puissant inhibiteur double de TNKS1 et TNKS2, avec des IC50 de 3 nM et 1 nM, respectivement, et a également une sélectivité 100 fois supérieure contre d'autres enzymes de la famille PARP, avec des IC50 de 2,0 μM, 0,5 μM et> 3 μM, pour PARP1 , PARP2 et PARP6, respectivement.

-
GC73919
AZD-9574-acid
AZD-9574-acid (70D), un inhibiteur du PPAR-1, peut être utilisé pour la synthèse du PROTAC (CAS 2923686-70-6).

-
GC73130
Basroparib
STP1002
Basroparib est un inhibiteur puissant de la Poly - ADP - ribose polymérase (PARP) avec une activité antitumorale.
-
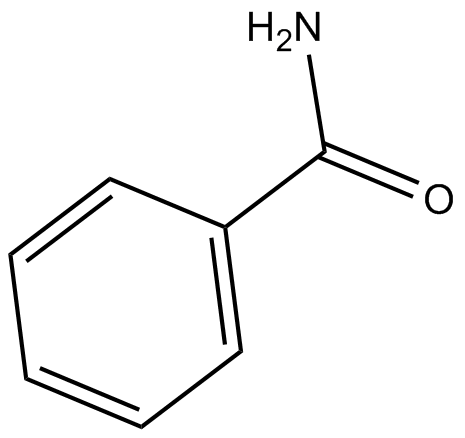
GC12844
Benzamide
Le benzamide (benzènecarboxamide) est un puissant inhibiteur de la poly(ADP-ribose) polymérase (PARP).
-
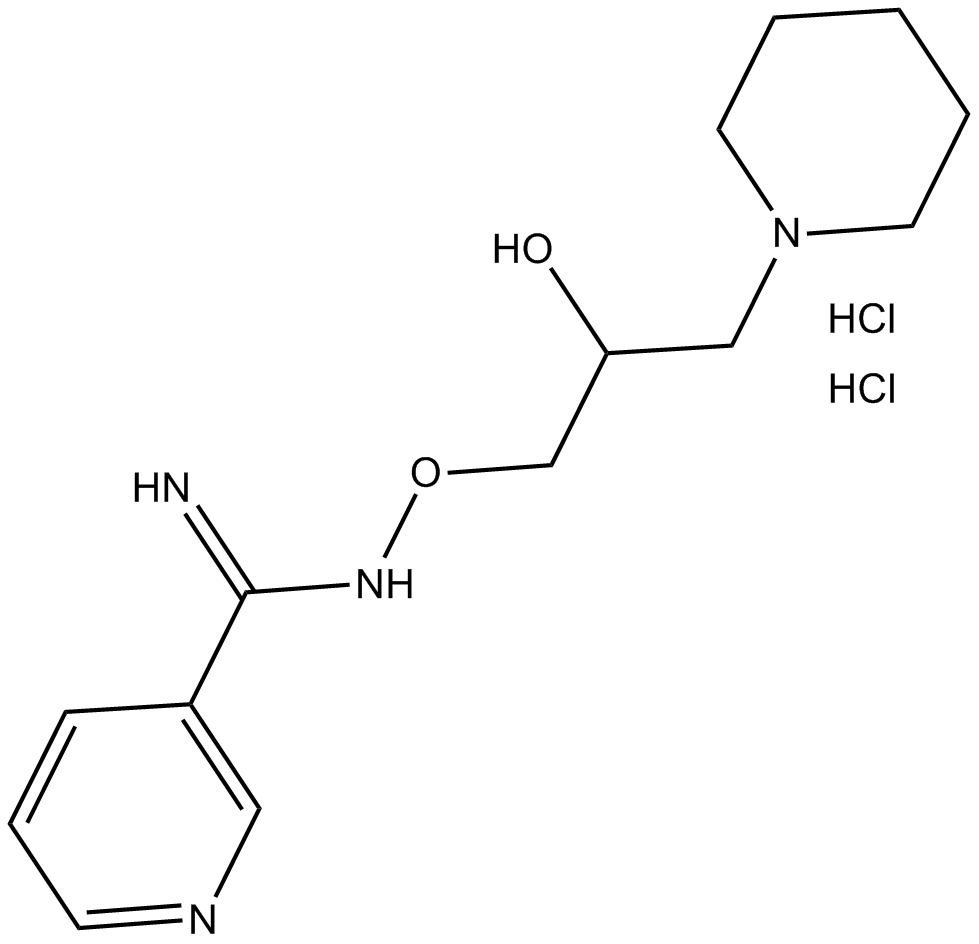
GC14380
BGP-15
BGP-15 est un inhibiteur de PARP, avec un IC50 et un Ki de 120 et 57 μM, respectivement.
-
GC35547
BR102375
BR102375 est un agoniste complet du récepteur activé par les proliférateurs de peroxysomes non-TZD (PPAR γ) pour le traitement du diabète de type 2, révèle une valeur EC50 de 0,28 μM et un rapport Amax de 98 %.

-
GC33223
BRCA1-IN-1
BRCA1-IN-1 est un nouvel inhibiteur de BRCA1 de type petite molécule avec IC50 et Ki de 0,53 μM et 0,71 μM, respectivement.

-
GC35550
BRCA1-IN-2
BRCA1-IN-2 (composé 15) est un inhibiteur de l'interaction protéine-protéine (PPI) perméable aux cellules pour BRCA1 avec une IC50 de 0,31 μM et un Kd de 0,3 μM, qui présente des activités antitumorales via la perturbation de BRCA1 (BRCT)2 /interactions protéiques.
-
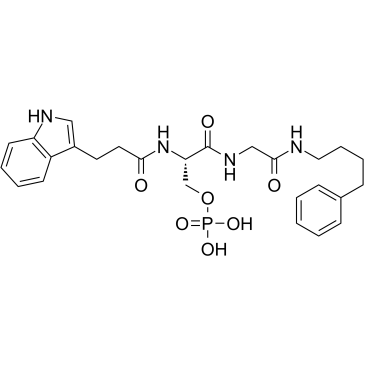
GC68940
DB008
DB008 est un inhibiteur sélectif efficace de PARP16 avec une valeur IC50 de 0,27 μM et contenant un réactif électrophile d'acrylamide. DB008 a une perméabilité membranaire et peut marquer sélectivement PARP16.
-
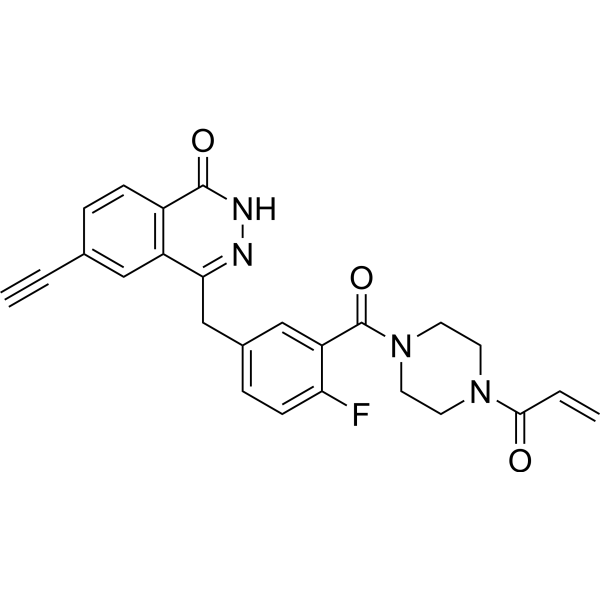
GC64209
E7016
GPI 21016
E7016 (GPI 21016) est un inhibiteur de PARP disponible par voie orale. E7016 peut améliorer la radiosensibilité des cellules tumorales in vitro et in vivo grÂce À l'inhibition de la réparation de l'ADN. E7016 agit comme un agent anticancéreux potentiel.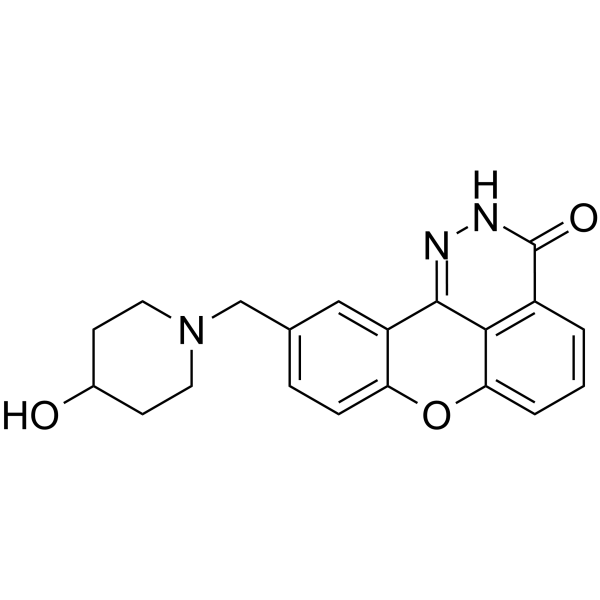
-
GC18172
E7449
E7449; 2X-121
E7449 est un puissant inhibiteur de PARP1 et PARP2 et inhibe également TNKS1 et TNKS2, avec des IC50 de 2,0, 1,0, ~50 et ~50 nM pour PARP1, PARP2, TNKS1 et TNKS2, respectivement, en utilisant 32P-NAD+ comme substrat.
-
GC13541
G007-LK
Tankyrase 1/2 Inhibitor VI
G007-LK est un inhibiteur puissant et sélectif de TNKS1 et TNKS2, avec des IC50 de 46 nM et 25 nM, respectivement.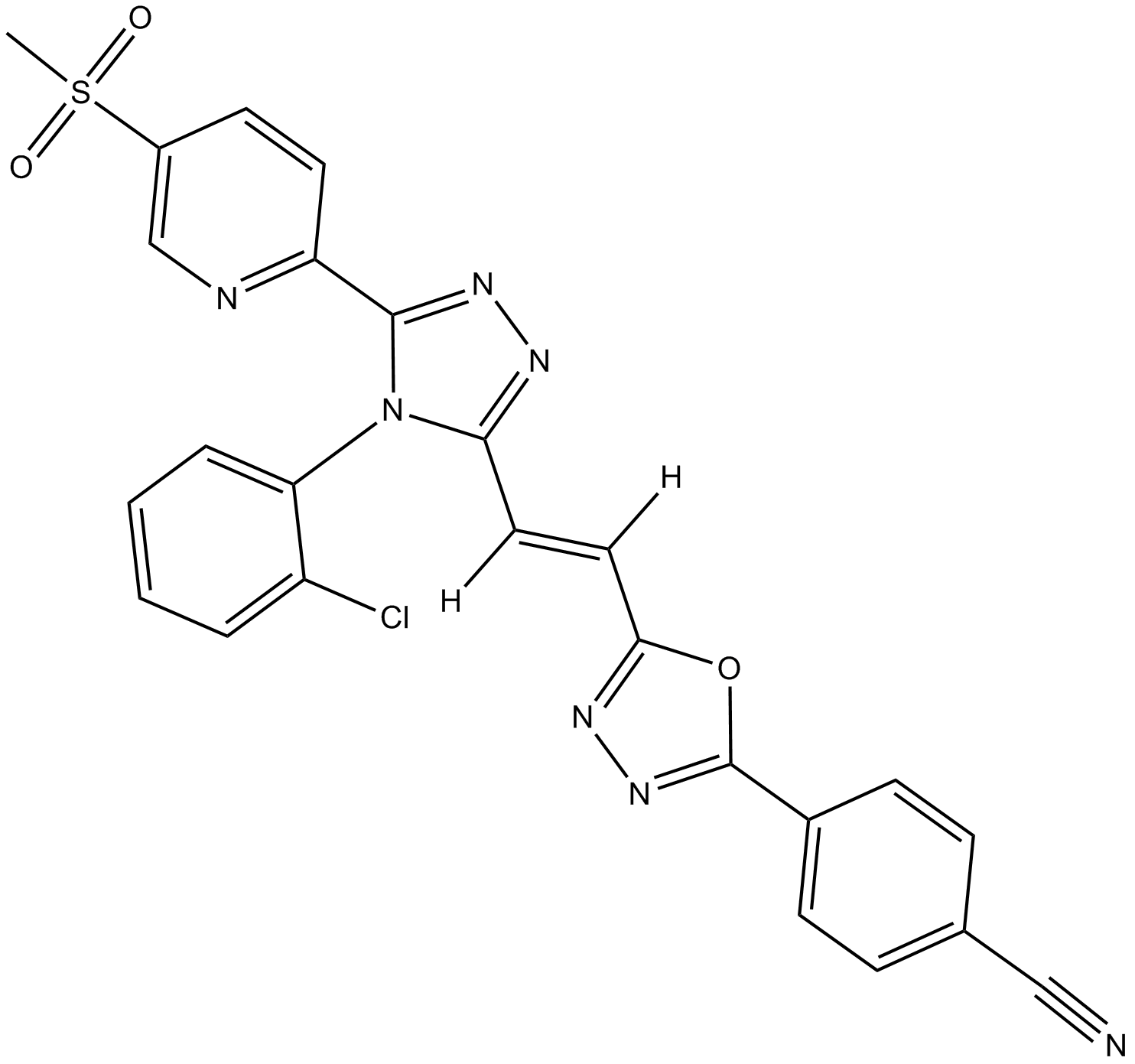
-
GC19542
GeA-69
GeA-69 est un inhibiteur allostérique sélectif de la poly-adénosine-diphosphate-ribose polymérase 14 (PARP14) ciblant le macrodomaine 2 (MD2), avec une valeur Kd de 2,1 µM. GeA-69 implique des mécanismes de réparation des dommages à l'ADN et empêche le recrutement de PARP14 MD2 sur les sites de dommages à l'ADN induits par laser.
-
GC38385
INO-1001

-
GC34195
K-756
Le K-756 est un inhibiteur direct et sélectif de la tankyrase (TNKS), qui inhibe l'activité d'ADP-ribosylation de TNKS1 et TNKS2 avec des IC50 de 31 et 36 nM, respectivement.

-
GC65907
KSQ-4279
USP1-IN-1
KSQ-4279 (USP1-IN-1, Formule I) est un inhibiteur de USP1 et PARP (extrait du brevet WO2021163530).
-
GC13419
ME0328
ME0328 est un inhibiteur ARTD3/PARP3 puissant et sélectif avec une IC50 de 0,89 ± 0,28 μM.

-
GC12756
MK-4827 hydrochloride
MK-4827 hydrochloride
Le chlorhydrate de MK-4827 (chlorhydrate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec des CI50 de 3,8 et 2,1 nM, respectivement. Le chlorhydrate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.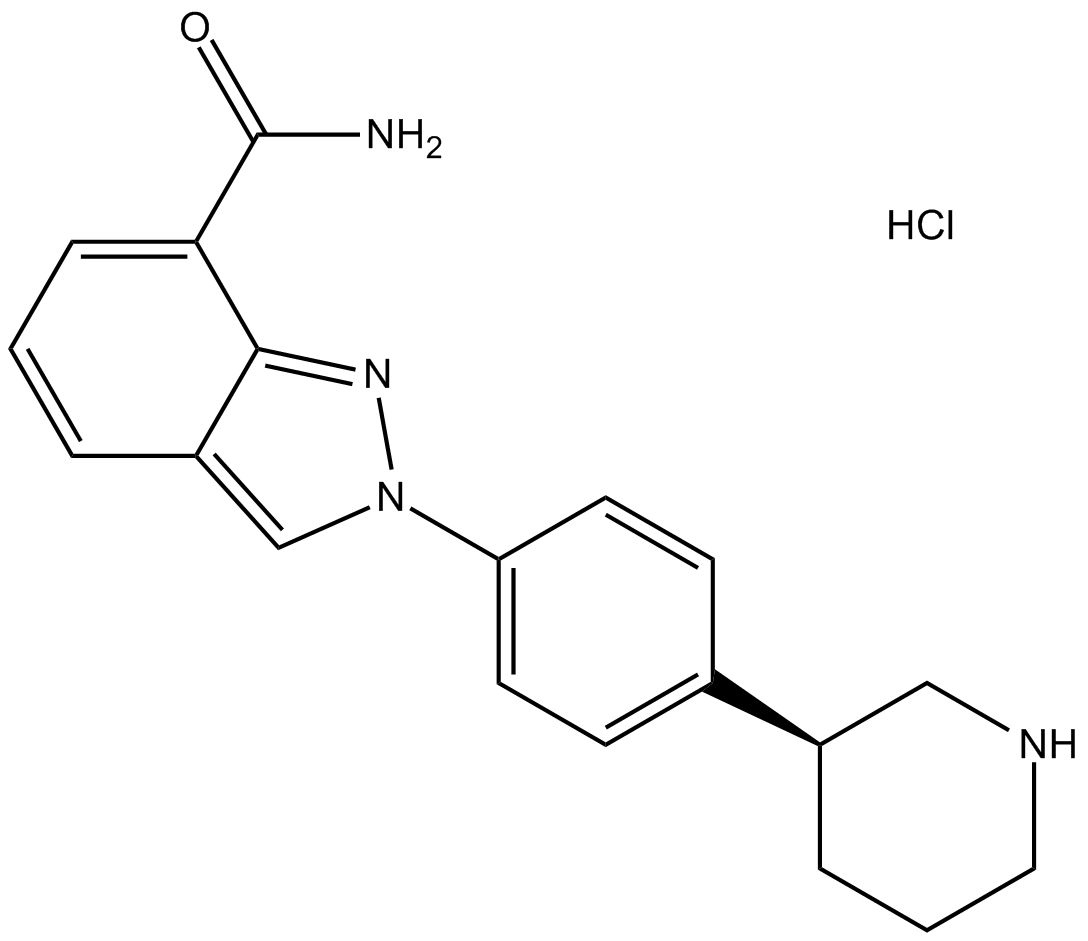
-
GC11537
MK-4827 tosylate
Niraparib tosylate
Le tosylate de MK-4827 (tosylate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec une IC50 de 3,8 et 2,1 nM, respectivement. Le tosylate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.
-
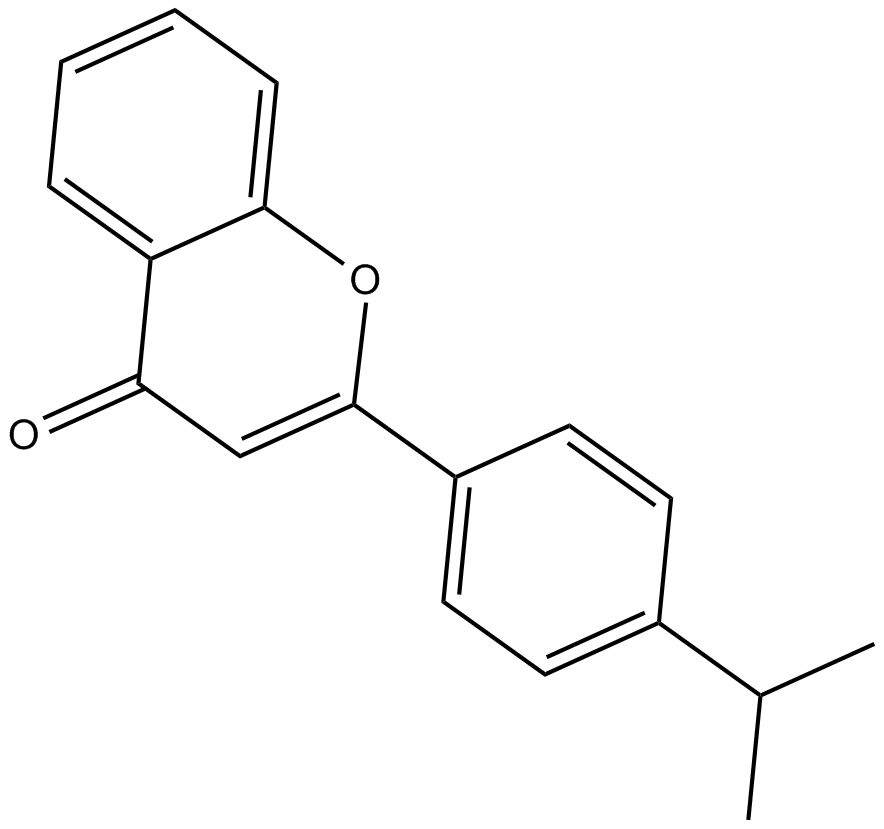
GC16914
MN 64
MN 64 est un puissant inhibiteur de la tankyrase 1, avec des CI50 de 6 nM, 72 nM, 19,1 μM et 39,4 μM pour TNKS1, TNKS2, ARTD1 et ARTD2, respectivement.
-
GC65202
Nesuparib
JPI-547/OCN-201
Le nésuparib est un puissant inhibiteur de la PARP.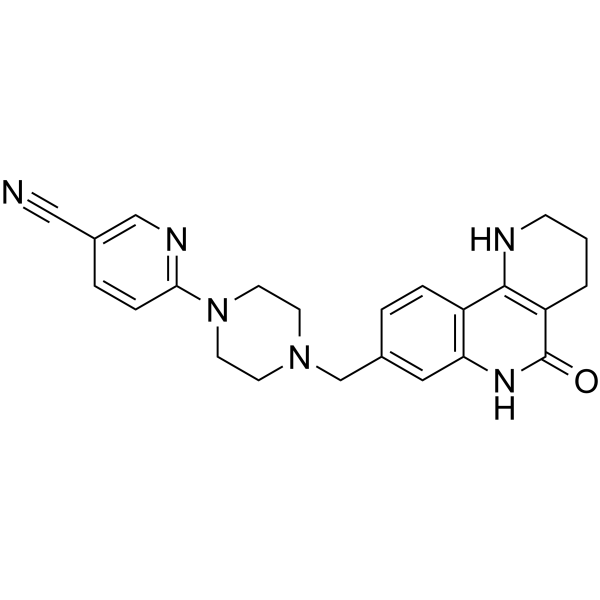
-
GC34120
Niraparib R-enantiomer (MK 4827 (R-enantiomer))
MK 4827 (R-enantiomer)
L'énantiomère R du niraparib (énantiomère MK-4827 R) est un excellent inhibiteur de PARP1 avec une IC50 de 2,4 nM.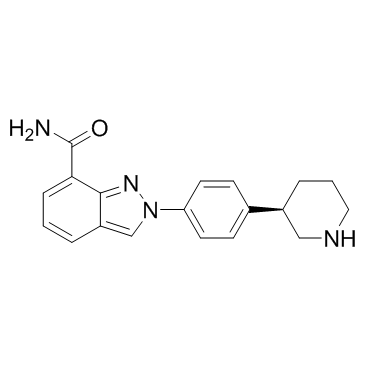
-
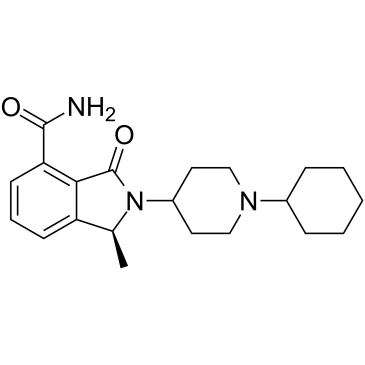
GC19264
NMS-P118
NMS-P118 est un puissant inhibiteur de PARP-1 disponible par voie orale et hautement sélectif pour le traitement du cancer.
-
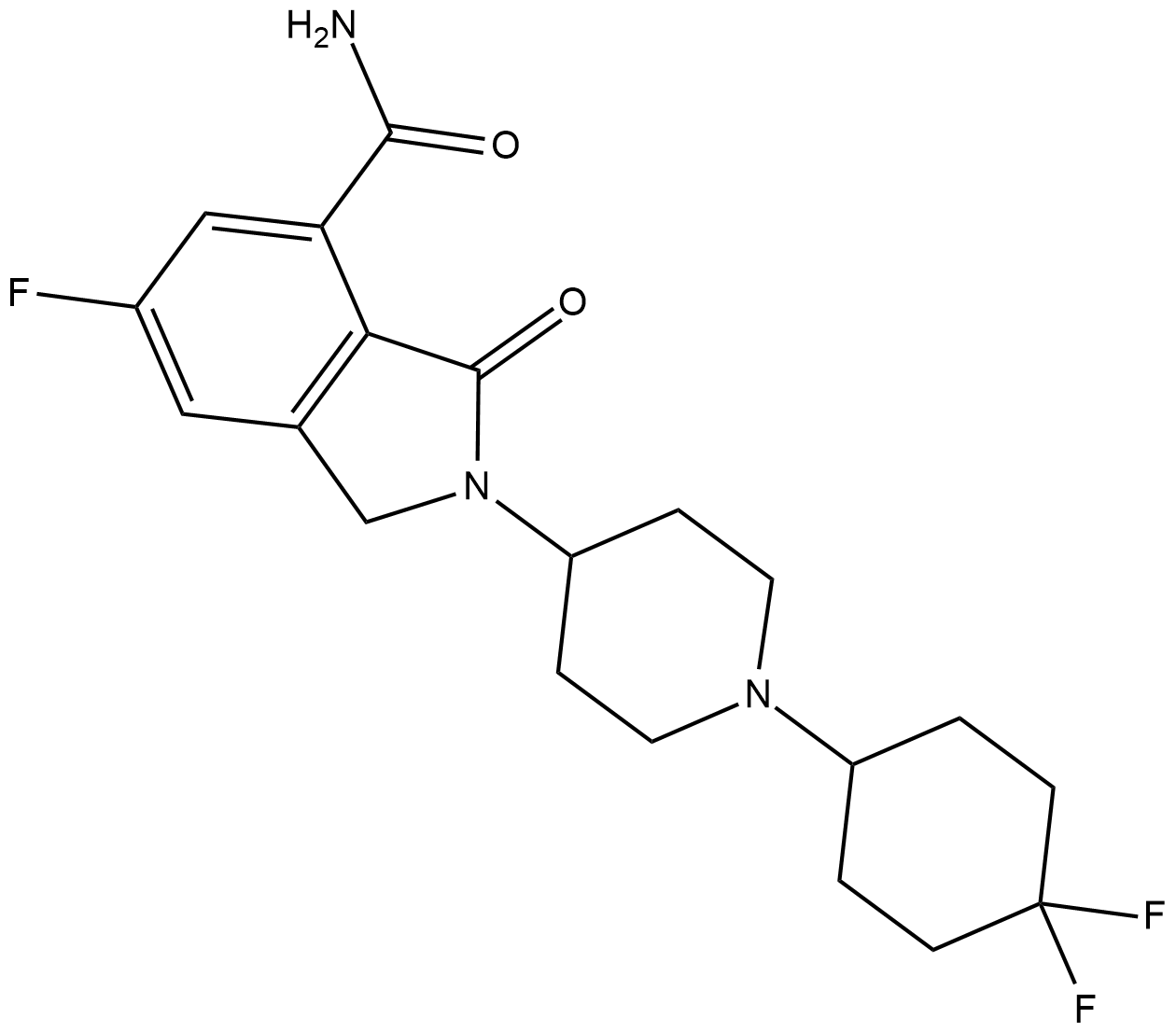
GC36751
NMS-P515
NMS-P515 est un inhibiteur de PARP-1 puissant, actif par voie orale et stéréospécifique, avec un Kd de 16 nM et une IC50 de 27 nM (dans les cellules Hela). Activité anti-tumorale.
-
GC17555
NVP-TNKS656
TNKS656
NVP-TNKS656 est un inhibiteur de TNKS2 très puissant, sélectif et actif par voie orale avec une IC50 de 6 nM et une sélectivité > 300 fois contre PARP1 et PARP2.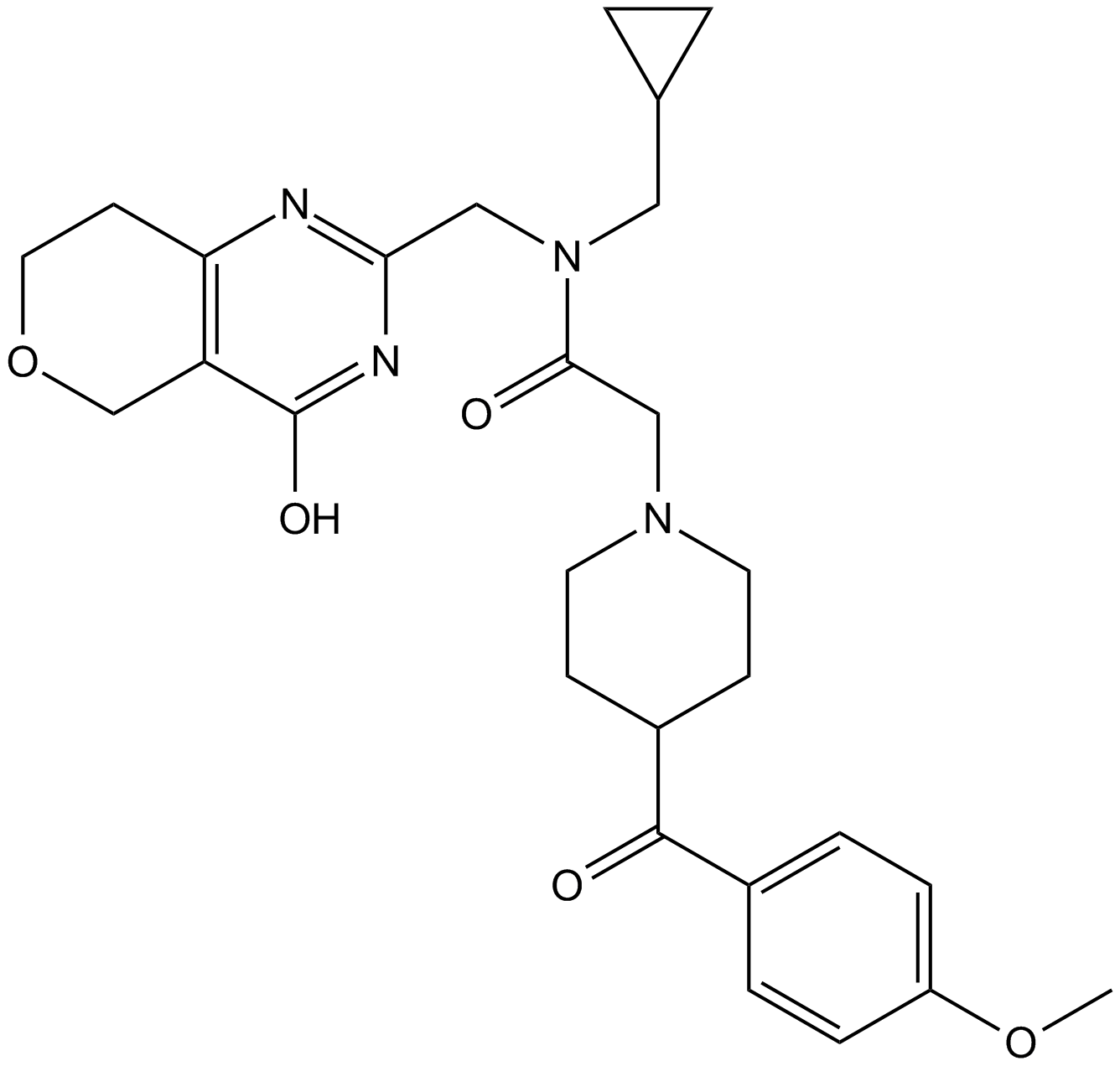
-
GC69618
Olaparib-d8
AZD2281-d8; KU0059436-d8
Olaparib-d8 est le deutérium de Olaparib (AZD2281). Olaparib est un inhibiteur oral efficace de PARP qui inhibe PARP-1 et PARP-2 avec des IC50 respectifs de 5 et 1 nM. Olaparib est également un activateur d'autophagie et de mitophagie.

-
GC67906
OM-153

-
GC73184
OUL232
OUL232 est un puissant inhibiteur des arts simples parp7, parp10, parp11, parp12, parp14 et parp15.

-
GC34071
Pamiparib (BGB-290)
BGB-290
Le pamiparib (BGB-290) (BGB-290) est un inhibiteur PARP actif par voie orale, puissant et hautement sélectif, avec des valeurs IC50 de 0,9 nM et 0,5 nM pour PARP1 et PARP2, respectivement. Le pamiparib (BGB-290) a un puissant piégeage PARP et une capacité À pénétrer dans le cerveau, et peut être utilisé pour la recherche de divers cancers, y compris la tumeur solide.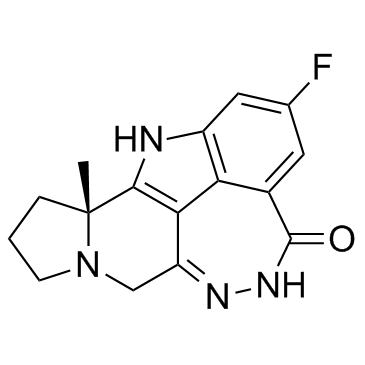
-
GC36855
Paris saponin VII
La saponine de Paris VII (Chonglou Saponin VII) est une saponine stéroÏdienne isolée des racines et des rhizomes de Trillium tschonoskii Maxim. L'apoptose induite par la saponine VII de Paris dans les cellules K562/ADR est associée À Akt/MAPK et À l'inhibition de la P-gp. La saponine de Paris VII atténue le potentiel de la membrane mitochondriale, augmente l'expression des protéines liées À l'apoptose, telles que Bax et le cytochrome c, et diminue les niveaux d'expression des protéines de Bcl-2, caspase-9, caspase-3, PARP-1 et p- Act. La saponine de Paris VII induit une autophagie robuste dans les cellules K562/ADR et fournit une base biochimique dans le traitement de la leucémie.

-
GC64579
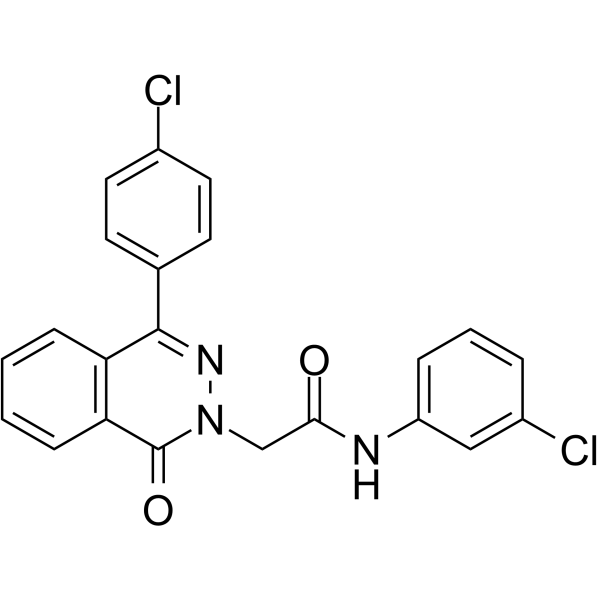
PARP-1-IN-2
PARP-1-IN-2 (composé 11g) est un puissant inhibiteur de PARP1 pénétré dans la BBB, avec une IC50 de 149 nM. PARP1-IN-2 montre une activité anti-proliférative significativement puissante contre la lignée cellulaire épithéliale d'adénocarcinome pulmonaire humain A549. PARP1-IN-2 peut induire l'apoptose des cellules A549.
-
GC73272
PARP-1-IN-3
PARP-1-IN-3 Le dérivé de benzamide est un puissant inhibiteur de PARP - 1 avec des IC50 de 0,25 nm pour PARP - 1 et de 2,34 nm pour PARP - 2.

-
GC65927
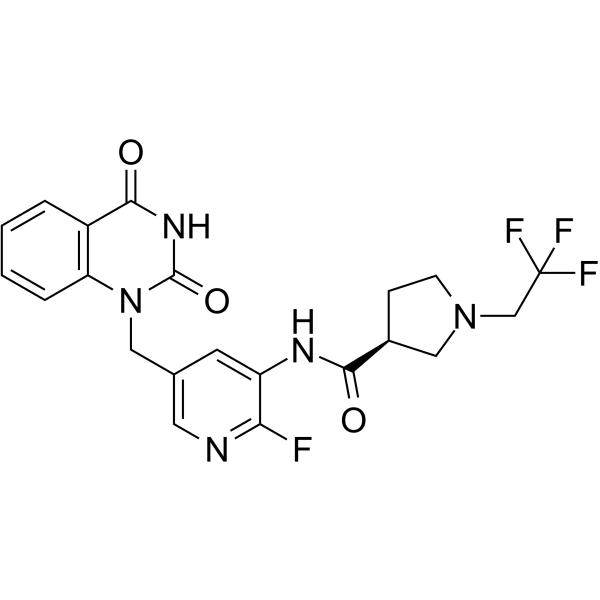
PARP-2-IN-1
PARP-2-IN-1 est un inhibiteur puissant et sélectif de PARP-2 avec une IC50 de 11,5 nM.
-
GC68006
PARP1-IN-11

-
GC74078
PARP1-IN-29
PARP1-IN-29 est un inhibiteur de la PARP-1 actif par voie orale avec une valeur ci50 de 6,3 nM.

-
GC69660
PARP1-IN-7
PARP1-IN-7 is an inhibitor of poly ADP-ribose polymerase-1 (PARP1), used as an anticancer agent.

-
GC64578
PARP1-IN-8
PARP1-IN-8 (composé 11c) est un puissant inhibiteur de PARP1 pénétré dans la BBB, avec une IC50 de 97 nM. PARP1-IN-8 montre une activité anti-proliférative significativement puissante contre la lignée cellulaire épithéliale d'adénocarcinome pulmonaire humain A549.

-
GC69657
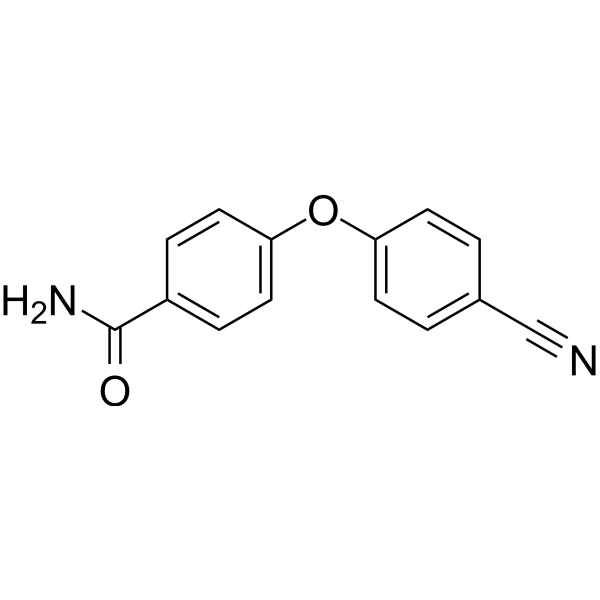
PARP10-IN-2
PARP10-IN-2 est un inhibiteur efficace de la PARP10, une mono-ADP-ribosyltransférase, avec une IC50 de 3,64 μM pour la PARP10 humaine. Il inhibe également la PARP2 et la PARP15, avec des IC50 respectifs de 27 μM et 11 μM pour les versions humaines de ces enzymes.
-
GC69658
PARP10-IN-3
PARP10-IN-3 est un inhibiteur sélectif de la poly(ADP-ribose) polymérase 10 (PARP10), une enzyme de transfert d'ADP-ribose monoétique. Son IC50 pour PARP10 humaine est de 480 nM. PARP10-IN-3 inhibe également les PARP2 et PARP15, avec des IC50 respectifs de 1,7 µM pour l'homme.

-
GC68035
PARP10/15-IN-1

-
GC69655
PARP10/15-IN-2
PARP10/15-IN-2 (Composé 8h) est un inhibiteur doublement efficace de PARP10 et PARP15, avec des valeurs IC50 respectives de 0,15 µM et 0,37 µM. PARP10/15-IN-2 peut pénétrer dans les cellules et empêcher l'apoptose cellulaire.

-
GC69656
PARP10/15-IN-3
PARP10/15-IN-3 (Composé 8a) est un inhibiteur doublement efficace de PARP10 et PARP15, avec des valeurs IC50 respectives de 0,14 µM et 0,40 µM. PARP10/15-IN-3 peut pénétrer dans les cellules et empêcher l'apoptose cellulaire.

-
GC69659
PARP11 inhibitor ITK7
ITK7
L'inhibiteur de PARP11 ITK7 (ITK7) est un inhibiteur sélectif efficace de PARP11. L'ITK7 peut efficacement inhiber le PARP11 avec une valeur IC50 de 14 nM. L'ITK7 peut être utilisé pour des études de localisation cellulaire.

-
GC69661
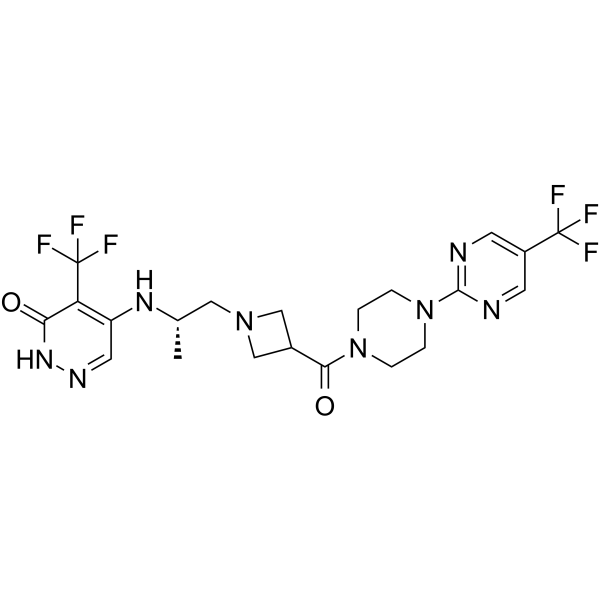
PARP7-IN-14
PARP7-IN-14 (I-1) est un inhibiteur sélectif efficace de PARP7 avec une valeur IC50 de 7,6 nM. PARP7-IN-14 possède une activité anticancéreuse.
-
GC73566
PARP7-IN-15
PARP7-IN-15 (composé 18) est un inhibiteur de parp7 avec une CI50 de 0,56 nm et une activité antitumorale.

-
GC73639
PARP7-IN-16
PARP7-IN-16 (composé 36) est un inhibiteur puissant, sélectif et actif par voie orale de PARP - 1 / 2 / 7 avec des CI50 de 0,94, 0,87 et 0,21 nm, respectivement.

-
GC73640
PARP7-IN-16 free base
PARP7-IN-16 free base est la forme de base libre de PARP7-IN-16.

-
GC14251
Picolinamide
poly (ADP-ribose) synthetase inhibitor

-
GC73943
Polθ/PARP-IN-1
Polθ/PARP-IN-1 (composé 25d) est un puissant inhibiteur double de la polymérase d’adn theta (Polθ) et de PARP avec des valeurs de ci50 de 45,6, 5,4 nM, respectivement.

-
GC65147
PROTAC PARP1 degrader
Le dégradeur PROTAC PARP1 est un dégradeur PARP1 basé sur le ligand MDM2 E3. Il induit un clivage important de PARP1 et une mort cellulaire programmée. Le dégradeur PROTAC PARP1 À 10 μM À 24 h inhibe la lignée cellulaire MDA-MB-231 avec une IC50 de 6,12 μM.

-
GC69804
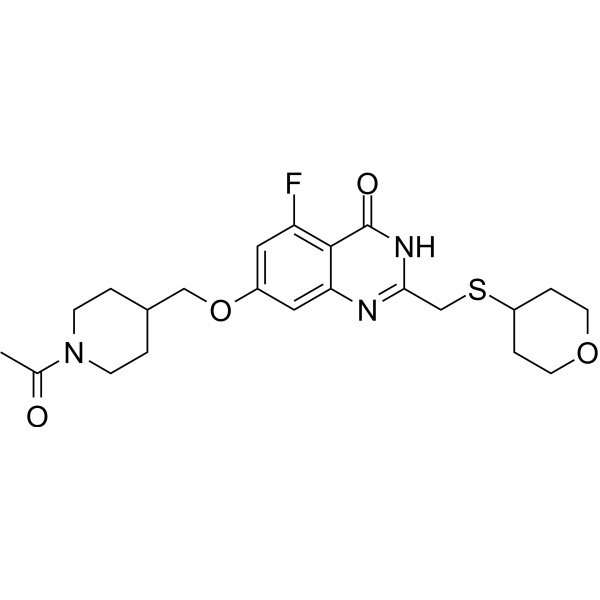
RBN-3143
RBN-3143 est un inhibiteur compétitif efficace de PARP14 catalysant la NAD+ avec une valeur IC50 de 4 nM. RBN-3143 inhibe l'ADP-ribosylation médiée par PARP14 et stabilise PARP14 dans les lignées cellulaires. RBN-3143 est utilisé pour la recherche sur l'inflammation pulmonaire.
-
GC72849
rel-PROTAC PARP1 degrader
rel-PROTAC PARP1 degrader est la configuration relative du dégradeur ROTAC PARP1.

-
GC19505
RK-287107
Le RK-287107 est un inhibiteur de tankyrase puissant et spécifique avec des IC50 de 14,3 et 10,6 nM pour la tankyrase-1 et la tankyrase-2, respectivement. Le RK-287107 bloque la croissance des cellules cancéreuses colorectales.

-
GC13249
Rucaparib (free base)
AG014447
Le rucaparib (base libre) (AG014699) est un inhibiteur puissant et actif par voie orale des protéines PARP (PARP-1, PARP-2 et PARP-3) avec un Ki de 1,4 nM pour PARP1. Le rucaparib (base libre) est un inhibiteur modeste de l'hexose-6-phosphate déshydrogénase (H6PD). Le rucaparib (base libre) a le potentiel pour la recherche sur le cancer de la prostate résistant À la castration (CRPC).
-
GC32793
Rucaparib Camsylate
Le monocamsylate de rucaparib (AG014699) est un inhibiteur puissant et actif par voie orale des protéines PARP (PARP-1, PARP-2 et PARP-3) avec un Ki de 1,4 nM pour PARP1. Rucaparib Camsylate est un inhibiteur modeste de l'hexose-6-phosphate déshydrogénase (H6PD). Rucaparib Camsylate a le potentiel pour la recherche sur le cancer de la prostate résistant À la castration (CRPC).

-
GC67962
Simmiparib

-
GC69907
SK-575
SK-575 est un inhibiteur de dégradation de PARP1 hautement efficace et spécifique, sous forme d'hybride ciblant la protéine hydrolysée (PROTAC), avec une IC50 de 2,30 nM. SK-575 peut efficacement inhiber la croissance des cellules cancéreuses portant des mutations BRCA1/2.

-
GC37728
Talazoparib tosylate
BMN 673ts
Talazoparib tosylate (BMN 673ts) est un inhibiteur de PARP1/2 puissant et actif par voie orale, qui est la forme de sel de tosylate du talazoparib. Talazoparib inhibe la PARP1 avec une valeur IC50 de 0,57 nM.
-
GC15041
Tankyrase Inhibitors (TNKS) 22
TNKS 22;TNKS22;TNKS-22
Tankyrase inhibitor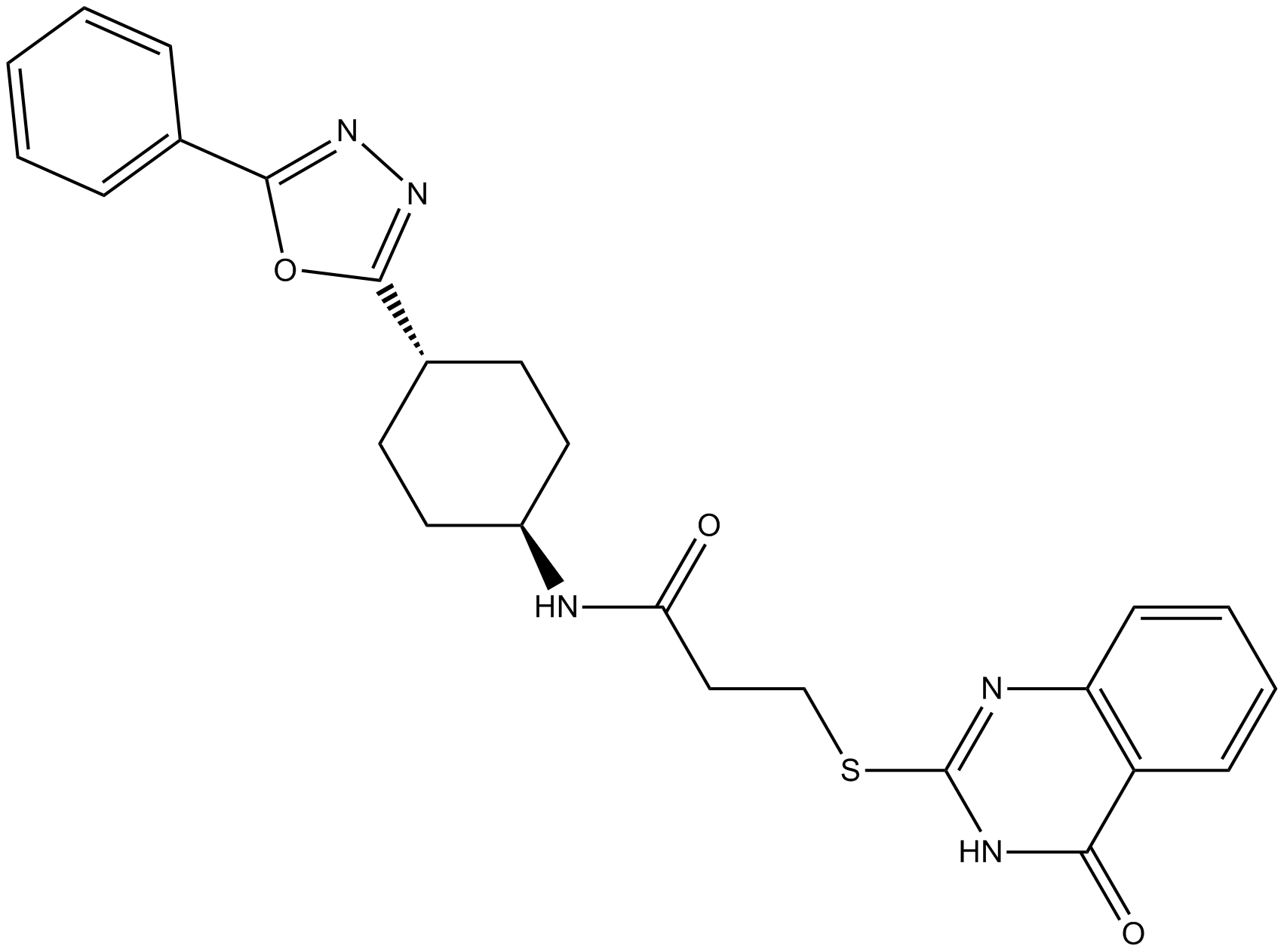
-
GC11548
Tankyrase Inhibitors (TNKS) 49
TNKS 49;TNKS49;TNKS-49
Tankyrase inhibitor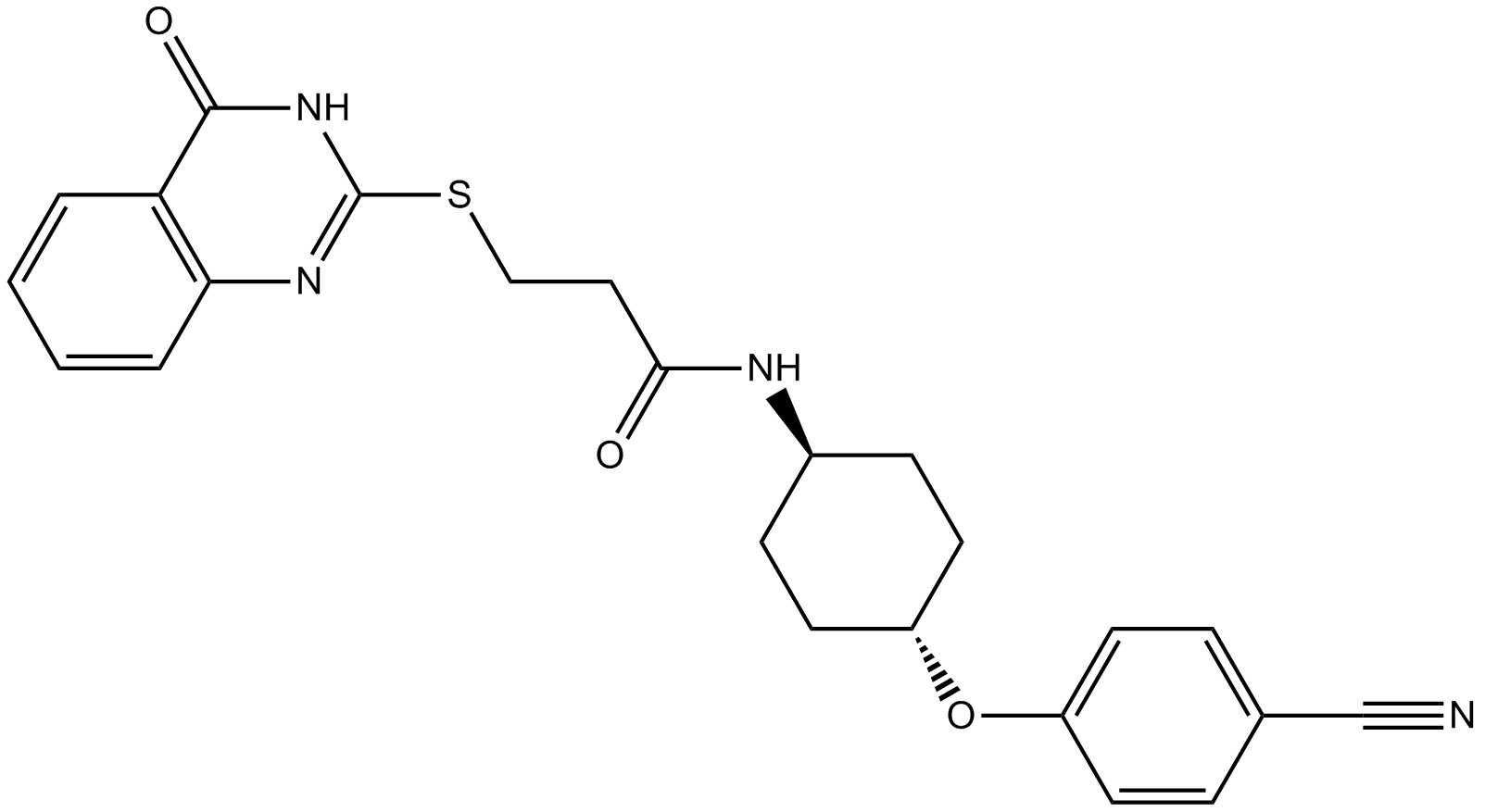
-
GC37735
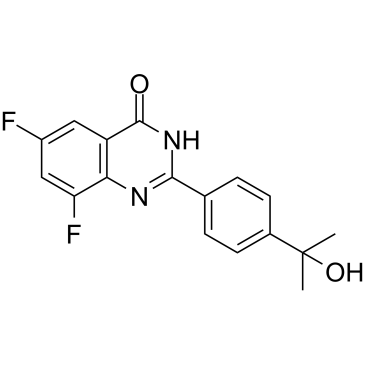
Tankyrase-IN-2
La tankyrase-IN-2 (composé 5k) est un inhibiteur de tankyrase puissant, sélectif et actif par voie orale (CI50 de 10, 7 et 710 nM pour TNKS1, TNKS2 ainsi que PARP1, respectivement). La tankyrase-IN-2 a un profil physicochimique et des propriétés pharmacocinétiques favorables modulant l'activité de la voie Wnt dans un modèle de xénogreffe colorectale.
-
GC71552
TNKS-2-IN-1
TNKS-2-IN-1 (composé 13G) est un inhibiteur de tnks - 2.

-
GC73633
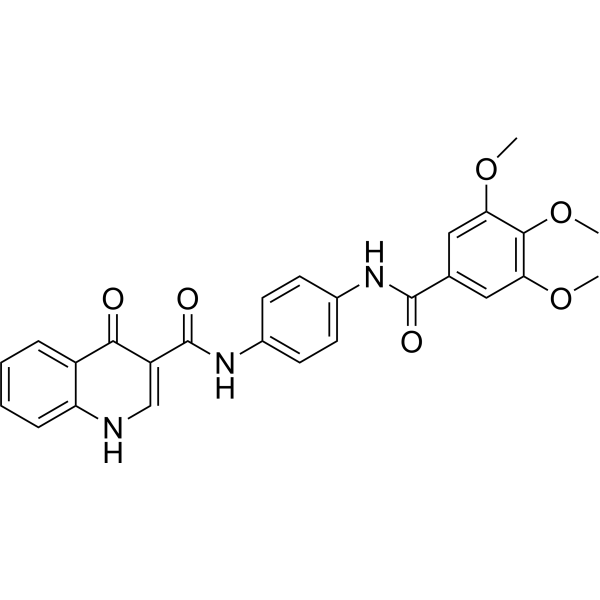
TNKS-2-IN-2
TNKS-2-IN-2 est un inhibiteur puissant et sélectif de TNKS2 avec une ci50 de 22 nM.
-
GC73760
Trilexium
TRX-E-009-1
Trilexium (TRX-E-009-1) est une troisième génération de benzopyran structurellement apparentée au TRX-E-002-1.
-
GC73136
VPC-70063
VPC-70063 est un puissant inhibiteur de myc - max avec une IC50 de 8,9 μm inhibant l'activité transcriptionnelle de myc - Max.

-
GC33358
WD2000-012547
WD2000-012547 est un inhibiteur sélectif de la poly(ADP-ribose)-polymérase (PARP-1) avec un pKi de 8,221.

-
GC12781
XAV-939
Le XAV-939 inhibe sélectivement la transcription médiée par la β-caténine.

-
GC91435
YCH1899
YCH1899 est un inhibiteur de la poly(ADP-ribose) polymérase 1 (PARP1) et PARP2 (IC50s = 1 Il est sélectif pour PARP1 et PARP2 par rapport à PARP3, -4, -5A, -5B, -6, -7, -10 et -12 (IC50s = 1-14.1 nM).




