Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(49)
- Aurora Kinase(59)
- DNA Methyltransferase(32)
- HDAC(187)
- Histone Acetyltransferases(70)
- Histone Demethylases(103)
- Histone Methyltransferase(205)
- HIF(94)
- JAK(157)
- MBT Domains(0)
- PARP(108)
- Pim(26)
- Protein Ser/Thr Phosphatases(26)
- RNA Polymerase(5)
- Sirtuin(77)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(22)
- Epigenetic Reader Domain(206)
- MicroRNA(20)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(20)
- DNA/RNA Demethylation(2)
- DNA/RNA Methylation(8)
- Histone Deacetylation(44)
- Histones/Histone Peptides(62)
- PHD Domains(1)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
- Readers(1)
- Small Interfering RNA (siRNA)(0)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
GC32929
MIR96-IN-1
MIR96-IN-1 cible le site Drosha dans le précurseur en épingle À cheveux miR-96 (miARN-96, microARN-96), inhibant sa biogenèse, déréprimant les cibles en aval et déclenchant l'apoptose dans les cellules cancéreuses du sein. MIR96-IN-1 se lie aux ARN avec des Kd de 1,3, 9,4, 3,4, 1,3 et 7,4 μM pour ARN1, ARN2, ARN3, ARN4 et ARN5, respectivement.

-
GC64487
Miravirsen
SPC-3649
Miravirsen (SPC-3649), un oligonucléotide antisens phosphorothioate modifié par un acide nucléique bloqué par β-d-oxy, inhibe la biogenèse de miR-122.
-
GC19248
Mivebresib
Mivebresib
Le mivébrésib (ABBV-075) est un inhibiteur de bromodomaine et de domaine extraterminal (BET) puissant et actif par voie orale. Le mivébrésib se lie À BRD4 avec un Ki de 1,5 nM.
-
GC17802
MK-4827
An orally bioavailable PARP1/2 inhibitor

-
GC12756
MK-4827 hydrochloride
MK-4827 hydrochloride
Le chlorhydrate de MK-4827 (chlorhydrate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec des CI50 de 3,8 et 2,1 nM, respectivement. Le chlorhydrate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.
-
GC17052
MK-4827 Racemate
selective inhibitor of PARP1/PARP2

-
GC11537
MK-4827 tosylate
Niraparib tosylate
Le tosylate de MK-4827 (tosylate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec une IC50 de 3,8 et 2,1 nM, respectivement. Le tosylate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.
-
GC17162
MK-5108 (VX-689)
VX-689
A potent inhibitor of Aurora kinases
-
GC19251
MK-8617
Le MK-8617 est un pan-inhibiteur actif par voie orale du facteur prolyl hydroxylase 1-3 induit par l'hypoxie (HIF PHD1-3) avec une IC50 de 1 nM pour PHD2.

-
GC10442
MK-8745
Le MK-8745 est un inhibiteur de la kinase Aurora A avec une IC50 de 0,6 nM.
-
GC14319
ML 228
CID-46742353
A HIF pathway activator
-
GC12557
ML324
CID-44143209
ML324 est un puissant inhibiteur de la déméthylase JMJD2 avec une activité antivirale. ML324 présente également une inhibition de l'histone déméthylase KDM4B, avec une IC50 de 4,9 μM. ML324 a une activité antivirale puissante contre l'infection par le virus de l'herpès simplex (HSV) et le cytomégalovirus humain (hCMV) via l'inhibition de l'expression du gène viral IE.
-
GC12208
MLN8054
An inhibitor of Aurora A kinase

-
GC12690
MLN8237 (Alisertib)
Alisertib
MLN8237 (Alisertib), en tant qu'inhibiteur sélectif de la kinase aurora A, disponible par voie orale et en cours d'investigation, est généralement utilisé pour le traitement des tumeurs solides et des maladies hématologiques malignes.
-
GC14652
MM-102
HMTase Inhibitor IX
MM-102 (HMTase Inhibitor IX) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki200 fois plus puissant que le peptide ARA.
-
GC36632
MM-102 TFA
HMTase Inhibitor IX TFA
Le MM-102 TFA (HMTase Inhibitor IX TFA) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki estimé 200 fois plus puissant que le peptide ARA.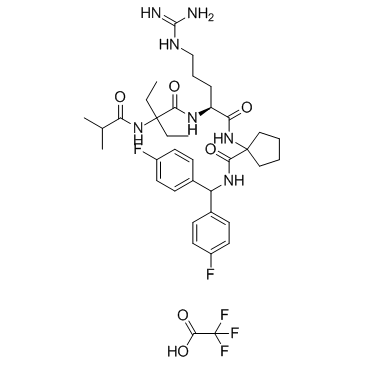
-
GC67758
MM-401 TFA

-
GC33278
MM-589
Le MM-589 est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.

-
GC65037
MM-589 TFA
Le MM-589 TFA est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.

-
GC72477
mmu-miR-1191a antagomir
mmu-miR-1191a antagomir sont des oligonucléotides modifiés chimiquement qui s’hybrident avec des miRNAs matures.
-
GC16914
MN 64
MN 64 est un puissant inhibiteur de la tankyrase 1, avec des CI50 de 6 nM, 72 nM, 19,1 μM et 39,4 μM pour TNKS1, TNKS2, ARTD1 et ARTD2, respectivement.
-
GC13055
Mocetinostat (MGCD0103, MG0103)
MGCD0103
An orally available HDAC inhibitor
-
GC34675
Molibresib besylate
GSK 525762C; I-BET 762 besylate
Le bésylate de molibresib (GSK 525762C; bésylate I-BET 762) est un inhibiteur du bromodomaine BET avec une IC50 de 32,5 À 42,5nM.
-
GC10046
Molidustat (BAY85-3934)
Molidustat
Molidustat (BAY85-3934) (BAY 85-3934) est un nouvel inhibiteur du facteur prolyl hydroxylase inductible par l'hypoxie (HIF-PH) avec des valeurs moyennes d'IC50 de 480 nM pour PHD1, 280 nM pour PHD2 et 450 nM pour PHD3.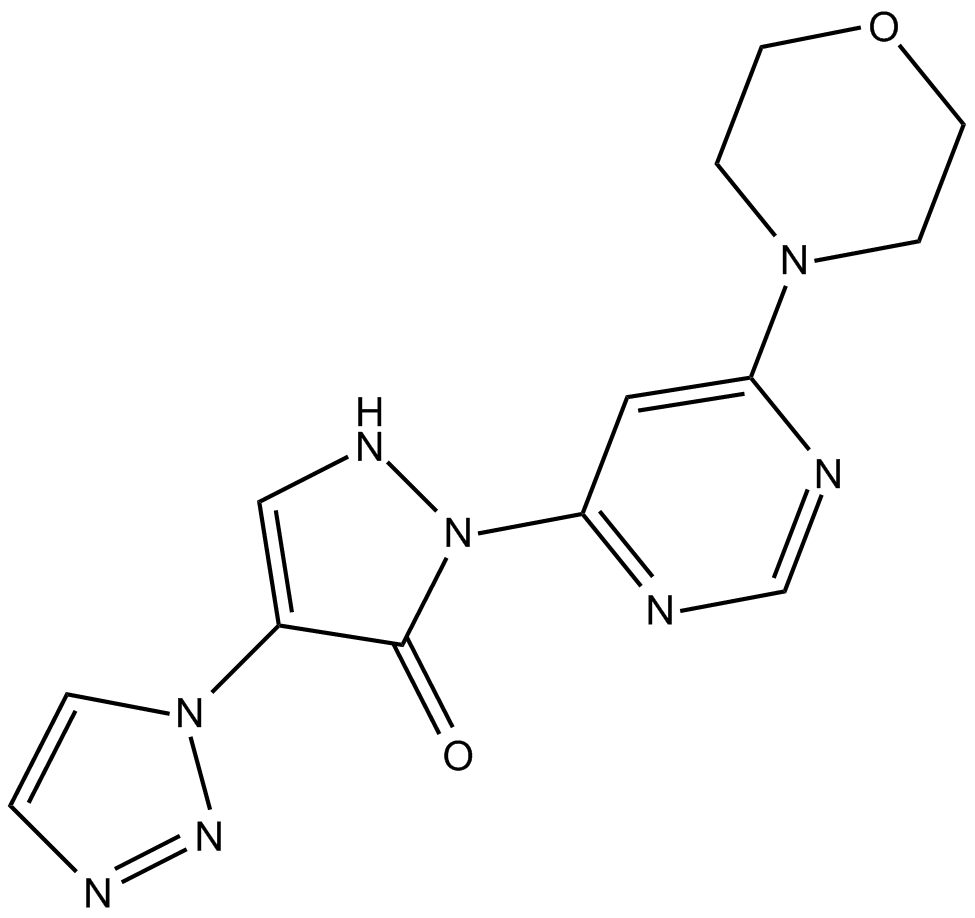
-
GC36646
Momelotinib Mesylate
CYT387 Mesylate
Le mésylate de momelotinib (CYT387 Mesylate) est un inhibiteur compétitif de l'ATP de JAK1/JAK2 avec une IC50 de 11 nM/18 nM, une sélectivité d'environ 10 fois par rapport À JAK3.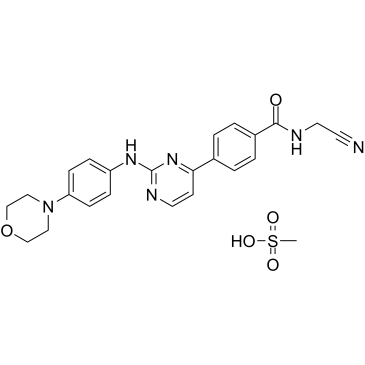
-
GC62406
Moracin O
Moracin O est un 2-arylbenzofurane isolé du Mori Cortex Radicis.

-
GC69488
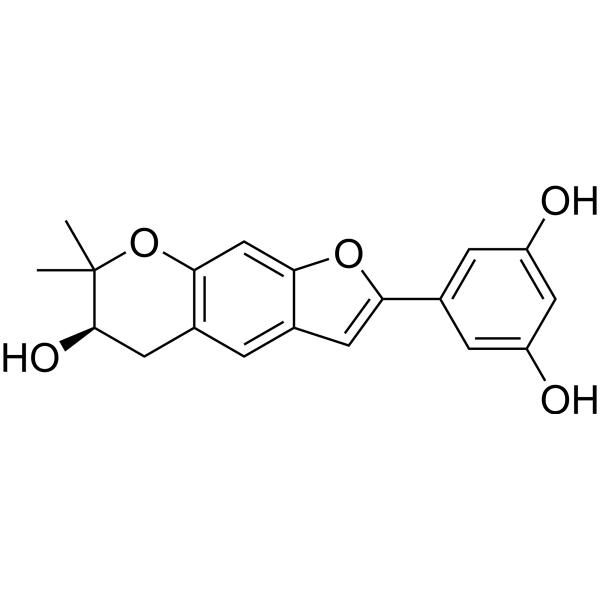
Moracin P
Moracin P est un 2-phénylbenzofurane isolé à partir de Mori Cortex Radicis. Moracin P présente une forte activité d'inhibition in vitro du facteur inducteur d'hypoxie (HIF-1). Moracin P réduit la production d'espèces réactives de l'oxygène (ROS) induite par le déprivation en oxygène et glucose (OGD). Moracin P a des effets neuroprotecteurs et anti-inflammatoires.
-
GC33371
MOZ-IN-2
MOZ-IN-2 est un inhibiteur de la protéine MOZ, membre des histones acétyltransférases, avec une IC50 de 125 μM.

-
GC64729
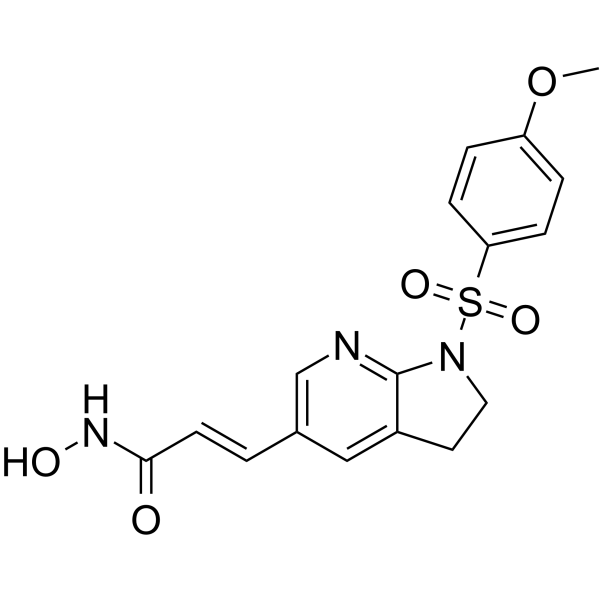
MPT0B390
MPT0B390 est un dérivé À base d'arylsulfonamide doté d'une puissante capacité inhibitrice d'HDAC. MPT0B390, inducteur de TIMP3, inhibe la croissance tumorale, les métastases et l'angiogenèse. MPT0B390 montre une activité antiproliférative contre la lignée cellulaire de cancer du cÔlon humain HCT116 avec le GI50 de 0,03 μM.
-
GC65965
MPT0E028
MPT0E028 est un inhibiteur HDAC actif et sélectif par voie orale avec des IC50 de 53,0 nM, 106,2 nM, 29,5 nM pour HDAC1, HDAC2 et HDAC6, respectivement. MPT0E028 réduit la viabilité des lymphomes À cellules B en induisant l'apoptose et possède une puissante capacité de ciblage direct Akt et réduit la phosphorylation d'Akt dans le lymphome À cellules B. MPT0E028 a une bonne activité anticancéreuse.

-
GC62308
MPT0G211
MPT0G211 est un puissant inhibiteur HDAC6 actif par voie orale et sélectif (IC50 = 0,291nM).

-
GC61468
MR837
MR837 est un inhibiteur de NSD2-PWWP1. MR837 peut se lier À la protéine 2 du domaine SET de liaison au récepteur nucléaire humain (domaine PWWP).

-
GC50576
MRK 740
Potent PRDM9 inhibitor

-
GC63558
MRTX-1719
MRTX-1719 est un inhibiteur sélectif de première classe puissant du complexe PRMT5/MTA, avec une IC50 de moins de 10 nM dans les cellules PRMT5/MTA MTAPDEL SDMA.

-
GC69499
MRTX-1719 hydrochloride
MRTX-1719 hydrochloride est un inhibiteur efficace, novateur et sélectif du complexe PRMT5/MTA. Son IC50 pour les cellules MTAPDEL SDMA de PRMT5/MTA est inférieur à 10 nM.

-
GC62715
MRTX9768
MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.

-
GC63080
MRTX9768 hydrochloride
Le chlorhydrate de MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.

-
GC40791
MS-1020
Le MS-1020 est un inhibiteur de JAK3 puissant et compétitif pour l'ATP. Le MS-1020 inhibe la signalisation JAK3/STAT et induit l'apoptose. MS-1020 favorise la mort cellulaire. Le MS-1020 diminue l'expression des niveaux de STAT3 phosphorylé par la tyrosine. MS-1020 a le potentiel pour la recherche de cancers hébergeant une signalisation JAK3 aberrante.

-
GC10099
MS023
MS023 est un inhibiteur puissant, sélectif et actif sur les cellules des méthyltransférases d'arginine de type I (PRMTs).

-
GC16432
MS023 (hydrochloride)
type I PRMTs inhibitor

-
GC36655
MS023 dihydrochloride
Le dichlorhydrate de MS023 est un inhibiteur puissant, sélectif et actif sur les cellules de l'inhibiteur de la protéine arginine méthyltransférase (PRMT) humaine de type I, avec des IC50 de 30, 119, 83, 4 et 5 nM pour PRMT1, PRMT3, PRMT4, PRMT6 et PRMT8, respectivement.

-
GC36656
MS049
MS049 est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 n'est pas toxique et n'affecte pas la croissance des cellules HEK293.

-
GC14240
MS049 (hydrochloride)
MS049 (chlorhydrate) est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 (chlorhydrate) réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 (chlorhydrate) n'est pas toxique et n'affecte pas la croissance des cellules HEK293.

-
GC36657
MS31
MS31 est un puissant inhibiteur de spindline 1 (SPIN1) de la protéine lecteur de méthyllysine de type fragment, hautement affin et sélectif. MS31 inhibe puissamment les interactions entre SPIN1 et H3K4me3 (IC50 = 77 nM, AlphaLISA ; 243 nM, FP). MS31 se lie sélectivement au domaine Tudor II de SPIN1 (Kd = 91 nM). MS31 inhibe puissamment la liaison des peptides contenant de la triméthyllysine À SPIN1. MS31 n'est pas toxique pour les cellules non tumorigènes.

-
GC39252
MS31 trihydrochloride
Le trichlorhydrate de MS31 est un puissant inhibiteur de spindline 1 (SPIN1) de la protéine lecteur de méthyllysine, hautement affin et sélectif. Le trichlorhydrate de MS31 inhibe puissamment les interactions entre SPIN1 et H3K4me3 (IC50 = 77 nM, AlphaLISA ; 243 nM, FP). Le trichlorhydrate de MS31 se lie sélectivement au domaine Tudor II de SPIN1 (Kd = 91 nM). Le trichlorhydrate de MS31 inhibe puissamment la liaison des peptides contenant de la triméthyllysine À SPIN1 et n'est pas toxique pour les cellules non tumorigènes.

-
GC44250
MS351
MS351 is an antagonist of chromobox 7 (CBX7) that acts by binding the CBX7 chromodomain.

-
GC12630
MS37452
MS37452 est un puissant inhibiteur de la liaison du chromodomaine CBX7 À H3K27me3, avec un Kd de 27,7 μM. MS37452 peut déréprimer la transcription du gène cible du complexe répressif polycomb p16/CDKN2A en déplaÇant la liaison de CBX7 au locus INK4A/ARF dans les cellules cancéreuses de la prostate.

-
GC64999
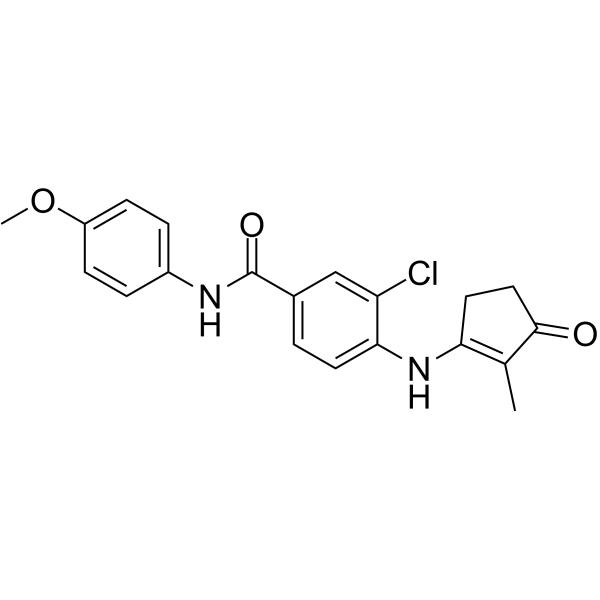
MS402
MS402 est un inhibiteur BET BrD sélectif de BD1 avec Kis de 77 nM, 718 nM, 110 nM, 200 nM, 83 nM et 240 nM pour BRD4(BD1), BRD4(BD2), BRD3(BD1), BRD3(BD2) , BRD2(BD1) et BRD2(BD2), respectivement.
-
GC31881
MS417 (GTPL7512)
GTPL7512
MS417 (GTPL7512) est un inhibiteur sélectif de BRD4 spécifique À BET, se lie À BRD4-BD1 et BRD4-BD2 avec des IC50 de 30, 46 nM et des Kd de 36,1, 25,4 nM, respectivement, avec une faible sélectivité À CBP BRD (IC50, 32,7 μ ;M).
-
GC13148
MS436
MS436 est une nouvelle classe d'inhibiteurs de bromodomaine, présente une affinité puissante d'un Ki = 30-50 nM estimé pour le BRD4 BrD1 et une sélectivité de 10 fois par rapport au BrD2.

-
GC38474
MS645
MS645 est un inhibiteur bivalent des bromodomaines BET (BrD) avec un Ki de 18,4 nM pour BRD4-BD1/BD2. MS645 contraint spatialement l'inhibition bivalente des BrD de BRD4, ce qui entraÎne une répression soutenue de l'activité transcriptionnelle de BRD4 dans les cellules tumorales solides.

-
GC64295
MS67
MS67 est un dégradeur puissant et sélectif de la protéine 5 à répétition WD40 (WDR5) avec une Kd de 63 nM. MS67 est inactif contre d'autres méthyltransférases, kinases, RCPG, canaux ioniques et transporteurs. MS67 montre des effets anticancéreux puissants.

-
GC71130
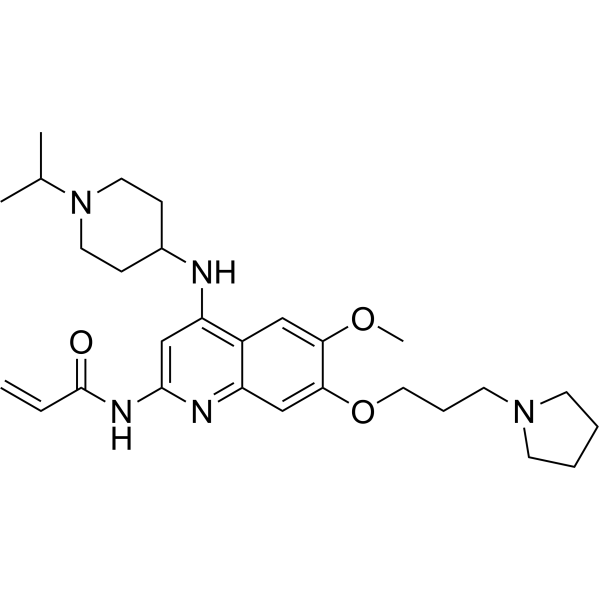
MS8511
MS8511 is a selective G9a/GLP covalent irreversible inhibitor by targeting a cysteine residue at the substrate binding site, with IC50 values of 100 nM (G9a) and 140 nM (GLP), and Kd values of 44 nM (G9a) and 46 nM (GLP).
-
GC69504
MS8815
MS8815 est un dégradeur PROTAC sélectif de la protéine homologue 2 de zeste (EZH2). MS8815 présente une activité inhibitrice sur EZH2 avec une valeur IC50 de 8,6 nM. MS8815 peut être utilisé dans la recherche sur le cancer du sein triple négatif (TNBC).

-
GC70682
MTL-CEBPA sodium
MTL-CEBPA sodium est la forme sodique de MTL - LCPE.
-
GC18729
MZ1
MZ1 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BRD4. MZ1 induit puissamment et rapidement une élimination réversible, durable et sélective de BRD4 sur BRD2 et BRD3. Kds de 382/120, 119/115 et 307/228 nM pour BRD4 BD1/2, BRD3 BD1/2 et BRD2 BD1/2, respectivement.

-
GC33102
MZP-54
MZP-54 est un PROTAC relié par des ligands pour von Hippel-Lindau et BRD3/4, avec un Kd de 4 nM pour Brd4BD2.

-
GC33363
MZP-55
MZP-55 est un PROTAC relié par des ligands pour von Hippel-Lindau et BRD3/4, avec un Kd de 8 nM pour Brd4BD2.

-
GC62154
N-Descyclopropanecarbaldehyde Olaparib
N-descyclopropanecarbaldéhyde L'olaparib est un analogue de l'olaparib contenant le fragment DOTA. Le N-descyclopropanecarbaldéhyde Olaparib est un ligand À base de CRBN pour la synthèse du nouveau double EGFR et PARP PROTAC, DP-C-4. N-Descyclopropanecarbaldéhyde L'olaparib peut être radiomarqué au F-18 ou au fluorophore pour la tomographie par émission de positrons (TEP) ou l'imagerie optique dans plusieurs types de tumeurs.

-
GC12458
N-Oxalylglycine
NOG
cell permeable inhibitor of α-ketoglutarate-dependent enzymes, including JMJD2A, JMJD2C, and JMJD2E.

-
GC40496
N6-Methyladenine
NSC 11580
N6-Methyladenine is a modified purine that is commonly found in genomes of prokaryotes, protists, and plants.
-
GC17676
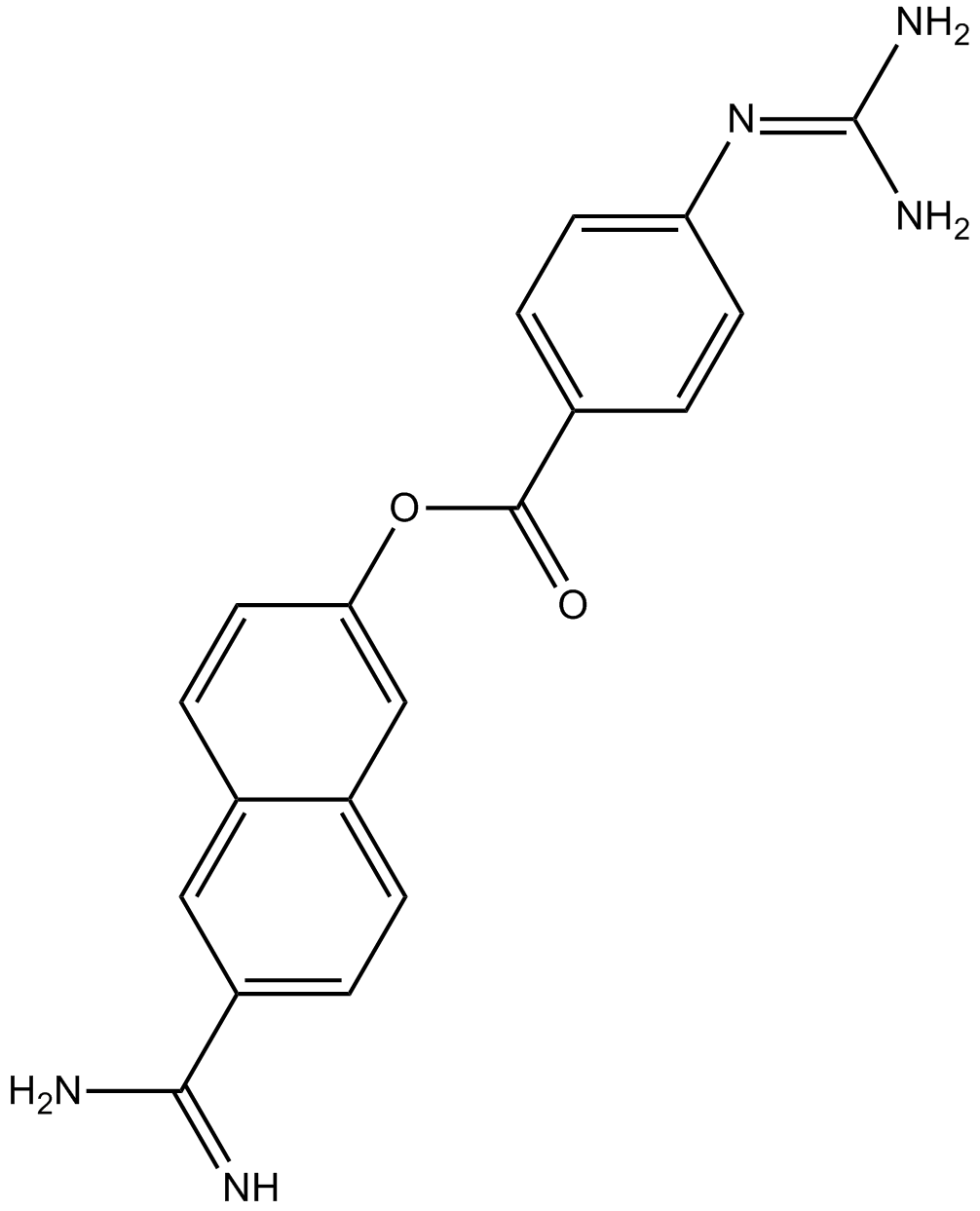
Nafamostat
Inhibiteur de la sérine protéase à large spectre, inhibiteur de la kallikréine.
-
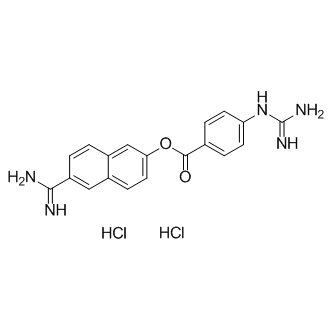
GC16290
Nafamostat hydrochloride
Le chlorhydrate de nafamostat, un inhibiteur synthétique de la sérine protéase, est un anticoagulant.
-
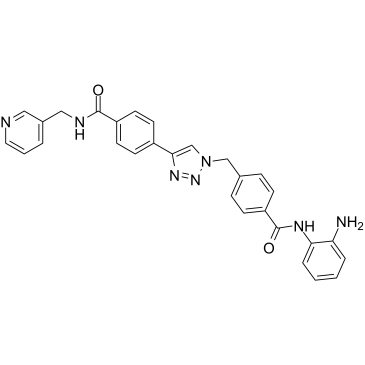
GC36690
Nampt-IN-3
Nampt-IN-3 (composé 35) inhibe simultanément la nicotinamide phosphoribosyltransférase (NAMPT) et l'HDAC avec des CI50 de 31 nM et 55 nM, respectivement. Nampt-IN-3 induit efficacement l'apoptose cellulaire et l'autophagie et conduit finalement À la mort cellulaire.
-
GC14562
Nanaomycin A
Nanafrocin, Nanafrocine, Nanafrocinum, Rosanomycin A
A bacterial metabolite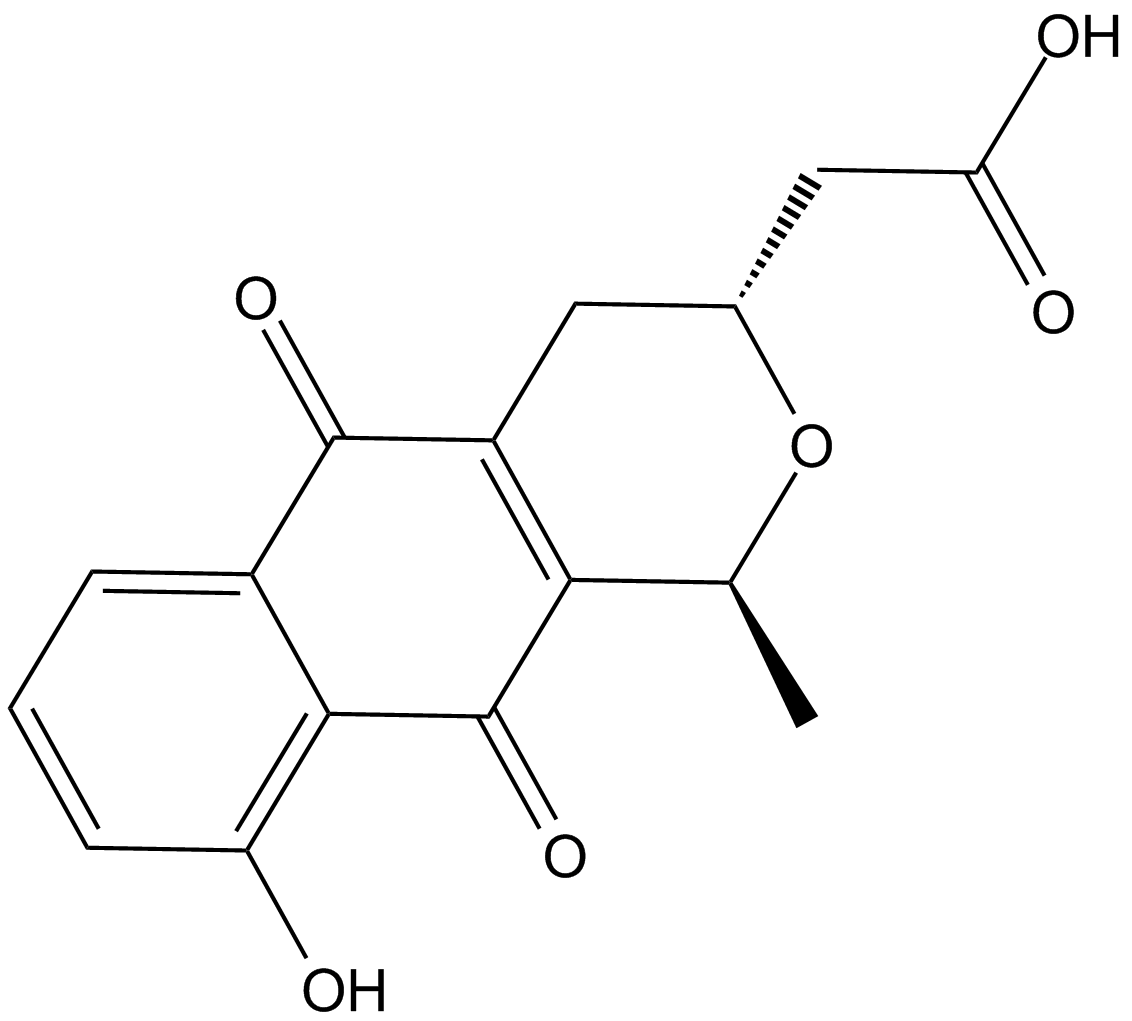
-
GC10243
Nanaomycin C
Amide of nanaomycin A

-
GC36691
Nanatinostat
CHR-3996
Le nanatinostat (CHR-3996) est un puissant inhibiteur de l'histone désacétylase (HDAC) sélectif de classe I et actif par voie orale avec une IC50 de 8 nM.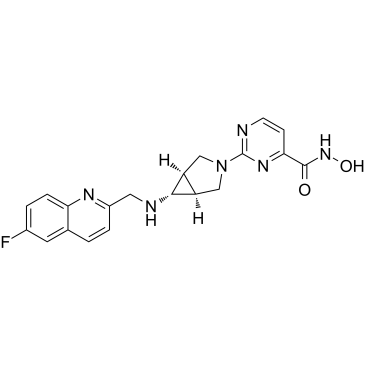
-
GC61841
Naphthol AS-E
Le naphtol AS-E est un inhibiteur puissant et perméable aux cellules de l'interaction KIX-KID. Le naphtol AS-E se lie directement au domaine KIX de CBP (Kd : 8,6 μM), bloque l'interaction entre le domaine KIX et le domaine KID de CREB avec une IC50 de 2,26 μM. Le naphtol AS-E peut être utilisé pour la recherche sur le cancer.

-
GC65138
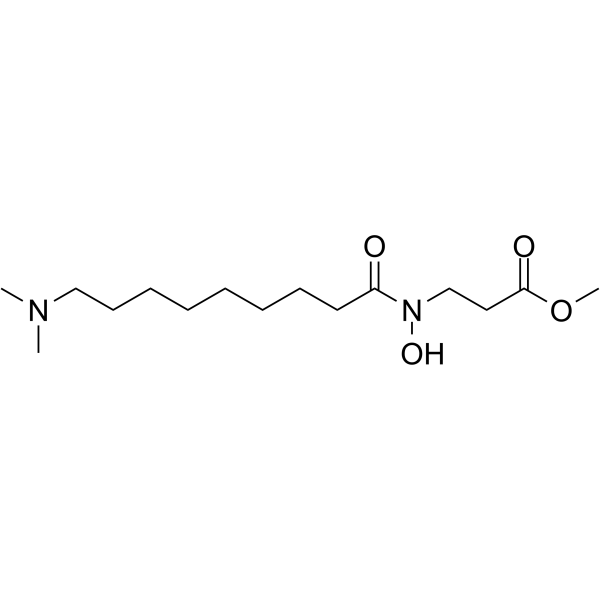
NCDM-32B
NCDM-32B est un inhibiteur puissant et sélectif de KDM4 qui altère la viabilité et transforme les phénotypes du cancer du sein.
-
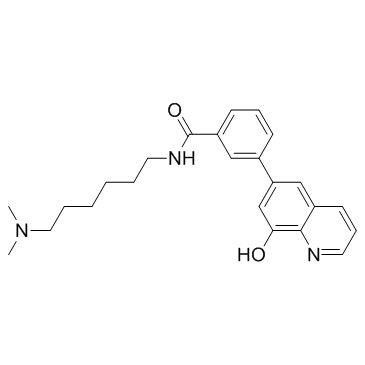
GC19410
NCGC00244536
NCGC00244536 est un puissant inhibiteur de KDM4B avec une IC50 de 10 nM.
-
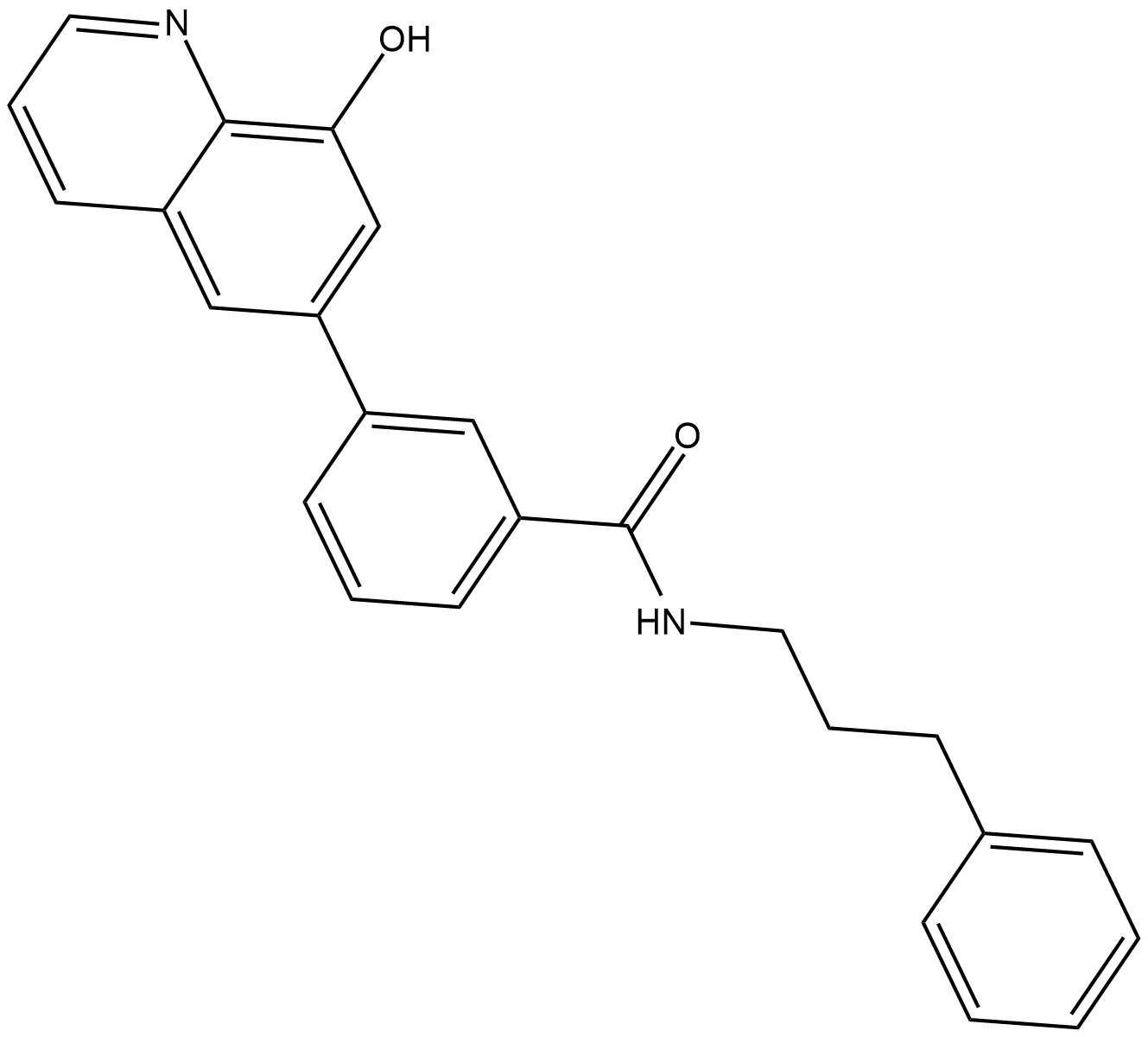
GC32896
NCGC00247743
NCGC00247743 est un inhibiteur de l'histone lysine déméthylase KDM4.
-
GC15536
NCH 51
NCH-51
An HDAC inhibitor
-
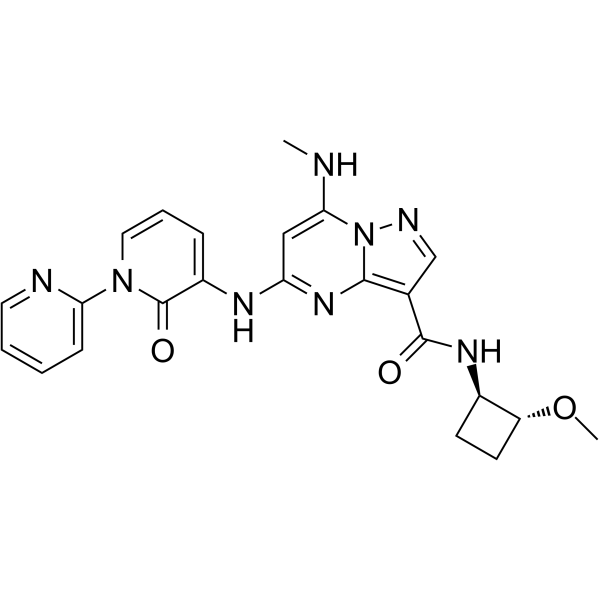
GC66329
NDI-034858
NDI-034858 est un inhibiteur de TYK2, cible le domaine TYK2 JH2 avec une constante de liaison Kd <200 pM.
-
GC65202
Nesuparib
JPI-547/OCN-201
Le nésuparib est un puissant inhibiteur de la PARP.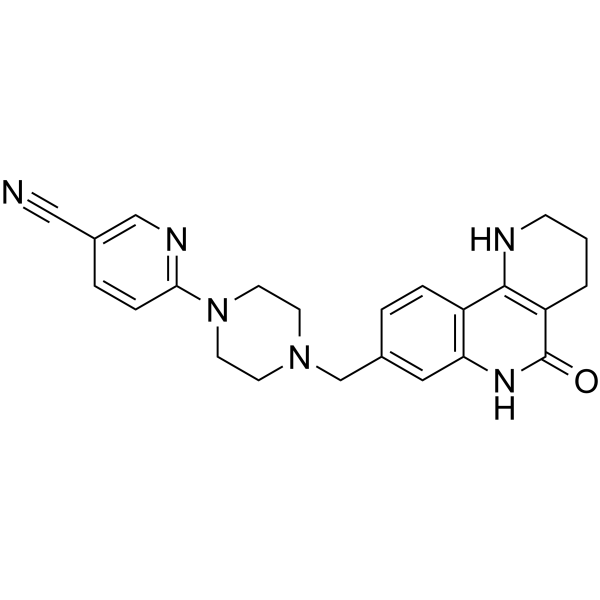
-
GC13764
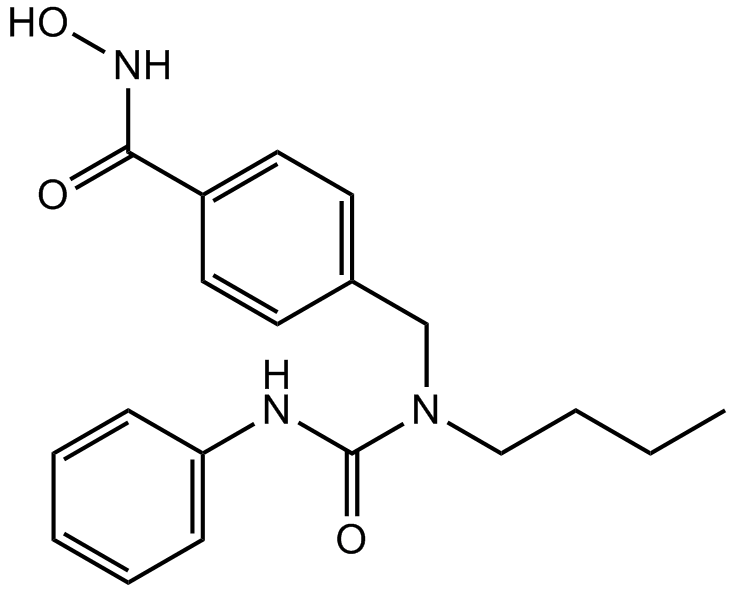
Nexturastat A
Le Nexturastat A est un puissant inhibiteur de HDAC6 avec une IC50 de 5 nM. Nexturastat A inhibe également HDAC10 et la protéine contenant le domaine métallo-β-lactamase2 (MBLAC2).
-
GC62620
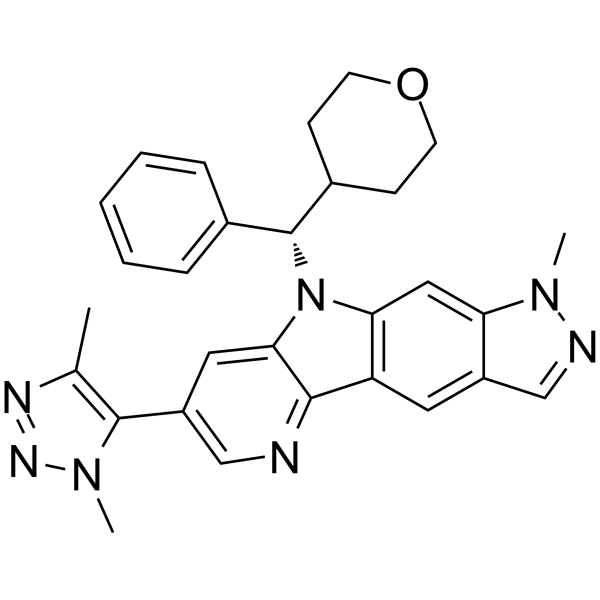
NHWD-870
NHWD-870 est un inhibiteur de bromodomaine puissant, actif par voie orale et sélectif de la famille BET et ne lie que les bromodomaines de BRD2, BRD3, BRD4 (IC50 = 2,7 nM) et BRDT. Le NHWD-870 a une puissante efficacité de suppression des tumeurs et supprime l'interaction cellules cancéreuses-macrophages. Le NHWD-870 augmente l'apoptose tumorale et inhibe la prolifération tumorale.
-
GC36734
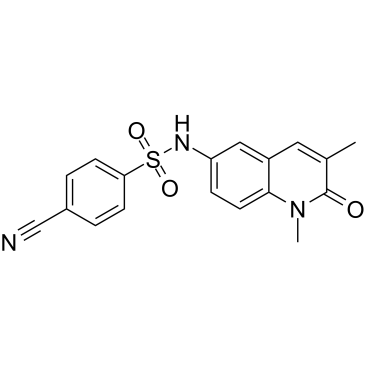
NI-42
NI-42 (composé 13-d), une sonde chimique structurellement orthogonale pour les BRPF, est un puissant inhibiteur biaisé de la BRD des BRPF (IC50 de BRPF1/2/3 = 7,9/48/260 nM; Kds de BRPF1 /2/3=40/210/940 nM) avec une excellente sélectivité sur les protéines BRD non classe IV.
-
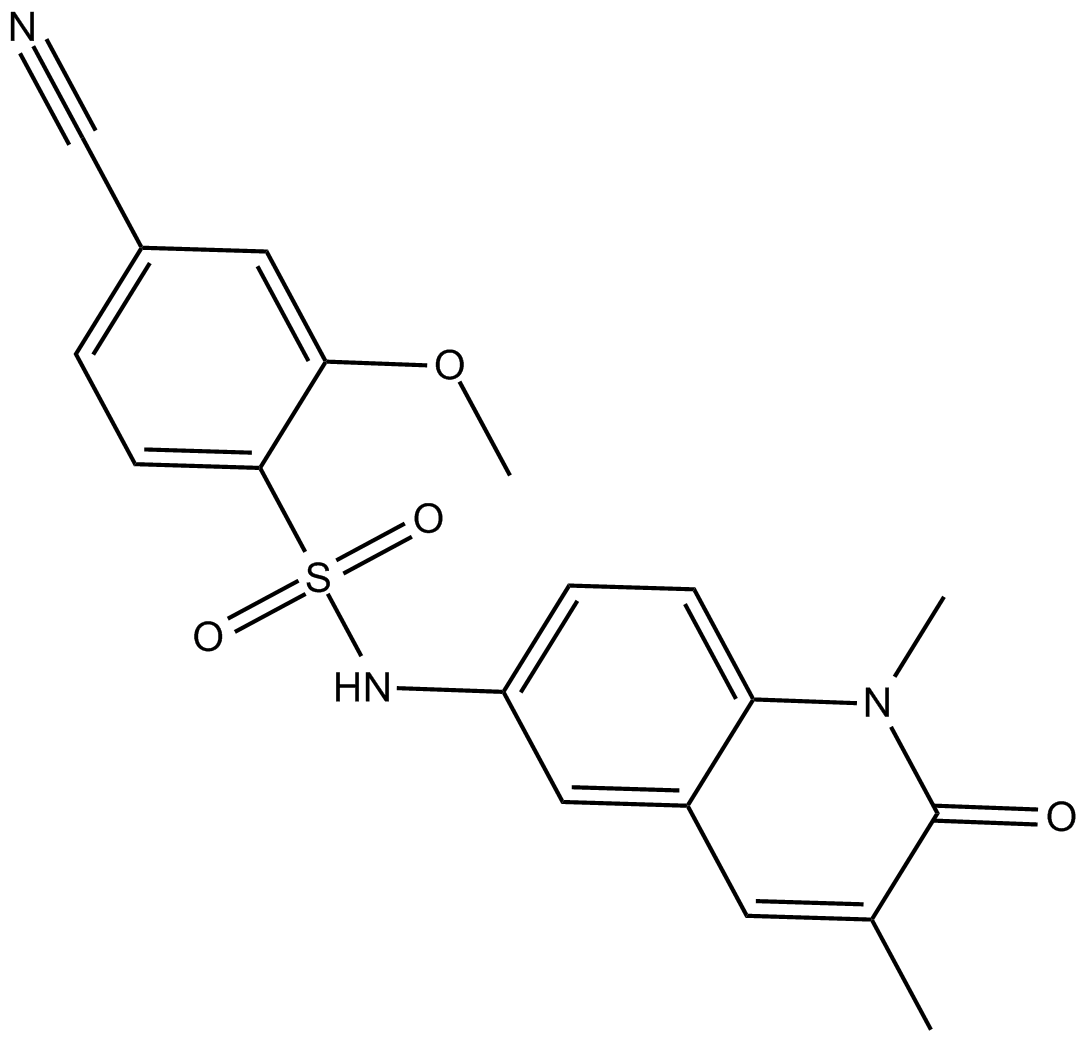
GC16763
NI-57
NI-57 est un inhibiteur de la famille de protéines bromodomaine et homéodomaine végétal contenant des doigts (BRPF), avec des IC50 de 3,1, 46 et 140 nM pour BRPF1, BRPF2 (BRD1) et BRPF3, respectivement.
-
GN10347
Nicotinamide (Vitamin B3)
Niacinamide, Nicotinic Acid Amide, NSC 13128, NSC 27452, Vitamin B3 Amide
Nicotinamide (Vitamin B3) est un dérivé amide de la vitamine B3 qui peut inhiber l'activité de la SIRT1 (IC50 : 50-180 μM) et de la SIRT2 (IC50 : 2 μM). Nicotinamide peut également augmenter les niveaux de NAD+, d'ATP et de ROS dans les cellules.
-
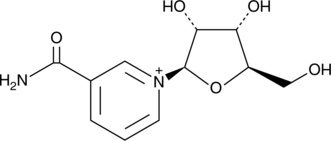
GC44401
Nicotinamide riboside
Le Nicotinamide riboside, une forme de vitamine B3 et un précurseur de NAD+, est converti en NAD+ biodisponible, via la kinase de nicotinamide riboside (NRK) et NMNAT, ou par l'action de la nucléoside phosphorylase et la récupération de NAM.
-
GC36738
Nicotinamide riboside chloride
Le chlorure de nicotinamide riboside, un précurseur de NAD+ actif par voie orale, augmente les niveaux de NAD+ et active SIRT1 et SIRT3.

-
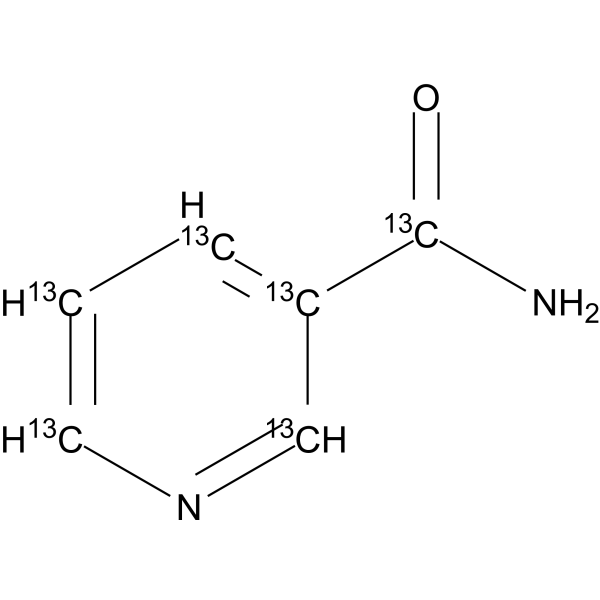
GC71615
Nicotinamide-13C6
Nicotinamide-13C6 est Nicotinamide marqué au 13C.
-
GC49270
Nicotinamide-d4
Niacinamide-d4, Nicotinic Acid Amide-d4, Vitamin B3 Amide-d4
Le nicotinamide-d4 (niacinamide-d4) est le nicotinamide marqué au deutérium.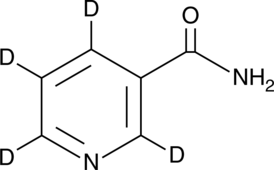
-
GC62248
NiCur
NiCur est un inhibiteur puissant et sélectif de l'histone acétyltransférase (HAT) de la CBP avec une valeur IC50 de 0,35 μμ. NiCur, qui bloque l'activité CBP HAT et régule À la baisse l'activation de p53 lors d'un stress génotoxique. NiCur peut être utilisé pour effectuer des études mécanistes sans affecter l'expression des protéines cibles.

-
GC71049
Nimucitinib
Nimucitinib est un inhibiteur de la Janus Kinase (Jak).
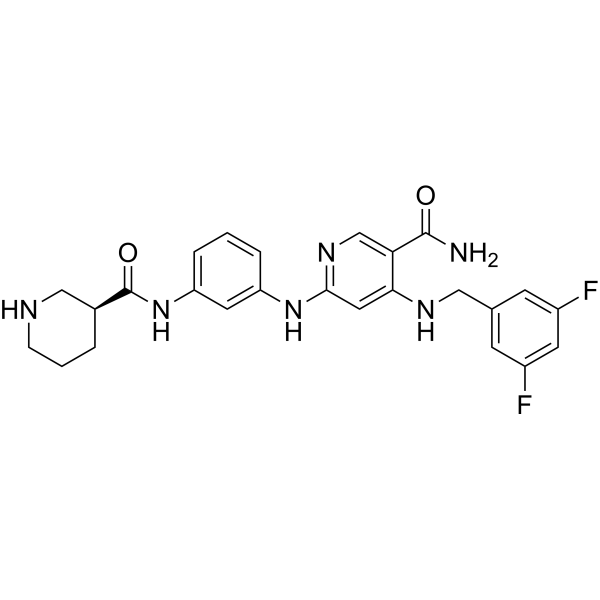
-
GC34120
Niraparib R-enantiomer (MK 4827 (R-enantiomer))
MK 4827 (R-enantiomer)
L'énantiomère R du niraparib (énantiomère MK-4827 R) est un excellent inhibiteur de PARP1 avec une IC50 de 2,4 nM.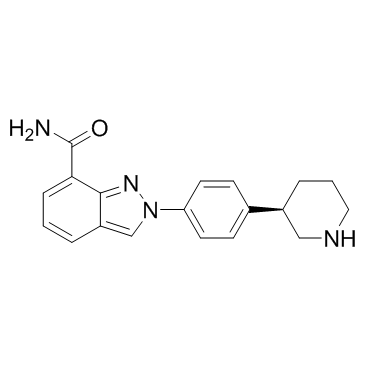
-
GC32964
NKL 22
Histone Deacetylase Inhibitor IV
NKL 22 (composé 4b) est un inhibiteur puissant et sélectif des histone désacétylases (HDAC), avec une IC50 de 199 et 69 nM pour HDAC1 et HDAC3, respectivement.
-
GC19264
NMS-P118
NMS-P118 est un puissant inhibiteur de PARP-1 disponible par voie orale et hautement sélectif pour le traitement du cancer.
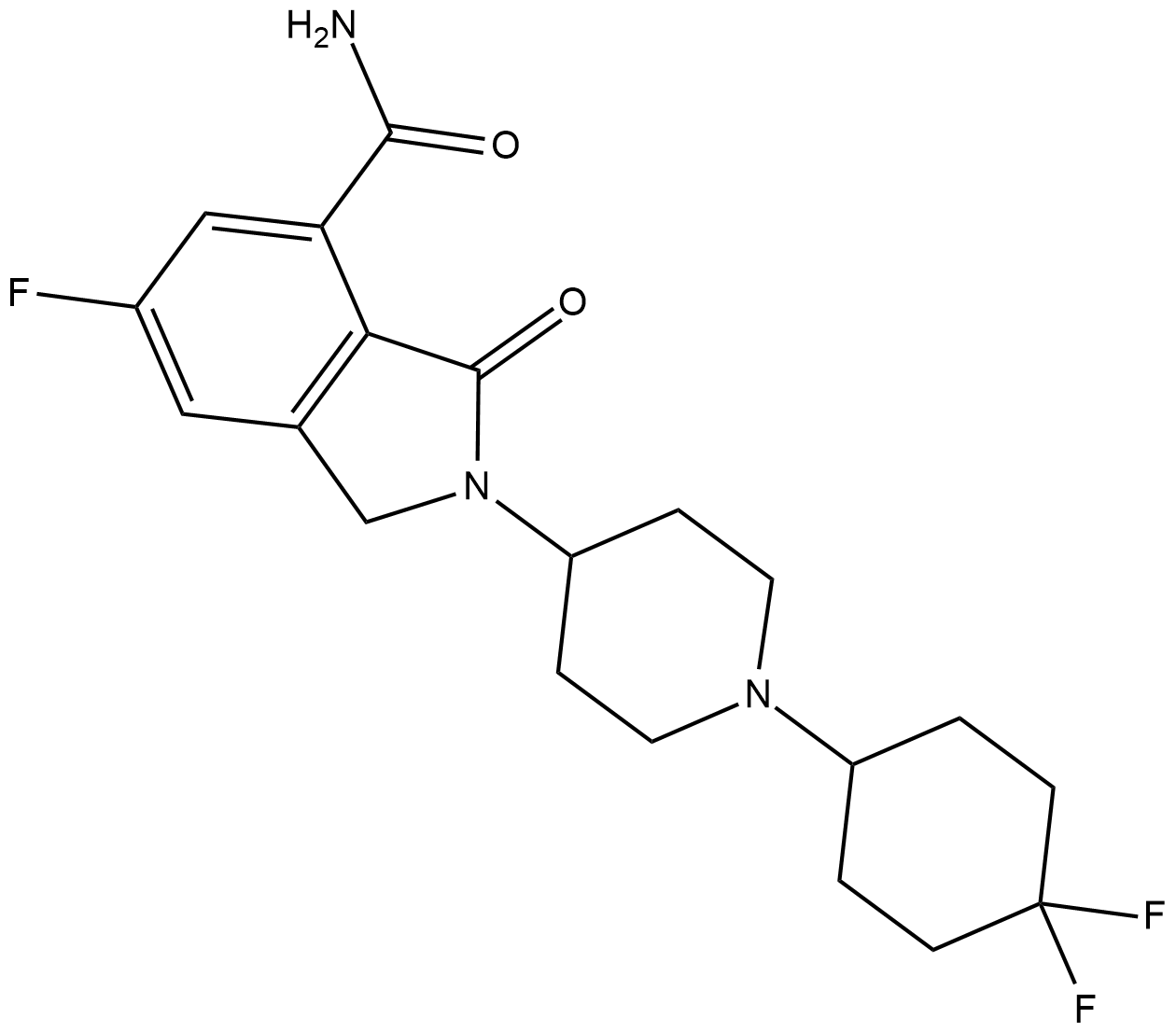
-
GC36751
NMS-P515
NMS-P515 est un inhibiteur de PARP-1 puissant, actif par voie orale et stéréospécifique, avec un Kd de 16 nM et une IC50 de 27 nM (dans les cellules Hela). Activité anti-tumorale.
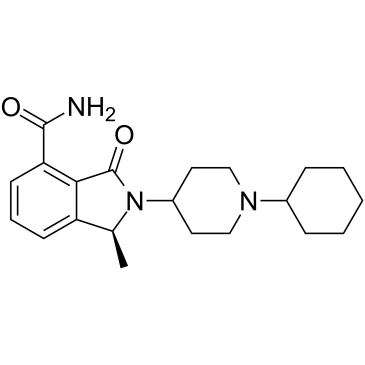
-
GC52166
NN-390
NN-390 est un inhibiteur HDAC6 puissant et sélectif, avec une IC50 de 9,8 nM. Le NN-390 traverse la barrière hémato-encéphalique (BBB). NN-390 montre un potentiel d'étude dans le groupe métastatique 3 MB (médulloblastome).
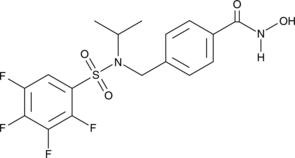
-
GC48460
NR-160
An inhibitor of HDAC6
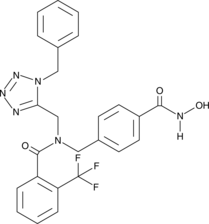
-
GC17762
NSC 33994
Selective inhibitor of JAK2 (IC50 = 60 nM). Displays no effect on Src and TYK2 tyrosine kinase activity at a concentration of 25 μM.

-
GC15040
NSC 3852
A tumor cell differentiating agent
-
GC50136
NSC 636819
NSC 636819 est un inhibiteur compétitif et sélectif de KDM4A/KDM4B. KDM4A/KDM4B sont des facteurs potentiels de progression du cancer de la prostate. NSC 636819 a le potentiel pour la recherche sur les maladies cancéreuses, en particulier le cancer de la prostate.
-
GC14875
NSC 663284
Cdc25 Phosphatase Inhibitor II, DA30031, SPS8I1
NSC 663284 (DA-3003-1) est un inhibiteur de la phosphatase À double spécificité Cdc25 puissant, perméable aux cellules et irréversible, a une IC50 pour Cdc25B2 de 0,21 μM. NSC 663284 présente une cinétique compétitive mixte contre Cdc25A, Cdc25B(2) et Cdc25C avec des valeurs Ki de 29, 95 et 89 nM, respectivement. NSC 663284 inhibe NSD2 (IC50 de 170 nM) par une interaction directe avec le domaine catalytique SET (Kd de 370 nM).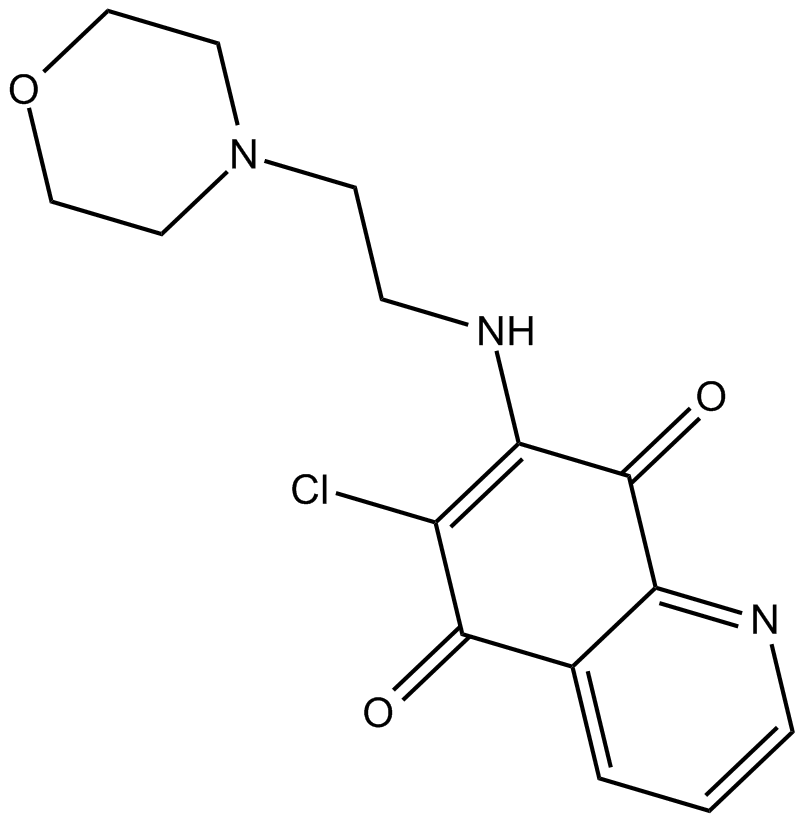
-
GC66049
NSC 694621
NSC 694621 est un puissant inhibiteur du PCAF, avec une IC50 de 5,71 μM (PCAF/H31-21). NSC 694621 présente une bonne activité d'inhibition de la prolifération des cellules cancéreuses.

-
GC69597
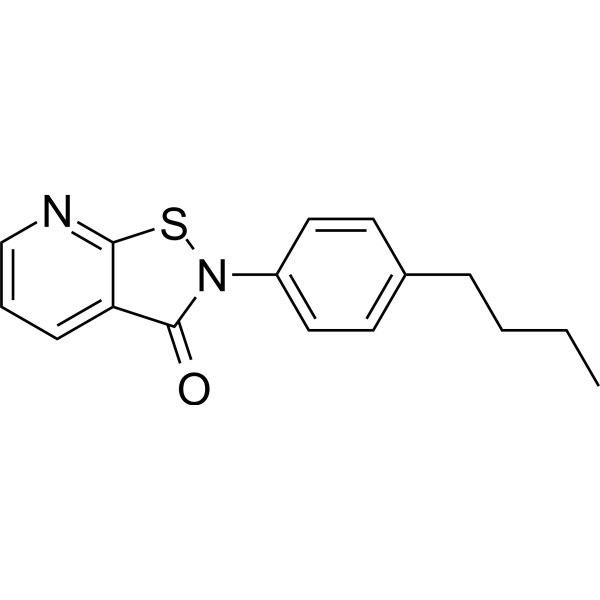
NSC 694623
NSC 694623 est un inhibiteur efficace de l'histone acétyltransférase (HAT), avec une IC50 de 15,9 μM pour le facteur associé à la HAT recombinante p300/CBP (PCAF). NSC 694623 a une activité anti-proliférative sur certaines cellules cancéreuses. NSC 694623 peut être utilisé dans la recherche contre le cancer.
-
GC49412
NSC 756093
NSC 756093 est un puissant inhibiteur de l'interaction GBP1:PIM1. NSC 756093 peut potentiellement inverser la résistance au paclitaxel. NSC 756093 peut être utilisé pour la recherche sur le cancer de l'ovaire.

-
GC11561
NSC 95397
Cdc25 Inhibitor IV, PTP Inhibitor XXIX
NSC 95397 est un puissant inhibiteur sélectif de la phosphatase À double spécificité Cdc25 (Ki = 32 nM (Cdc25A), 96 nM (Cdc25B), 40 nM (Cdc25C); IC50 = 22,3 nM (Cdc25A humain), 56,9 nM (Cdc25C humain), 125 nM (Cdc25B)). NSC 95397 inhibe la protéine kinase phosphatase-1 activée par les mitogènes (MKP-1), supprime la prolifération et induit l'apoptose dans les cellules cancéreuses du cÔlon par les voies MKP-1 et ERK1/2.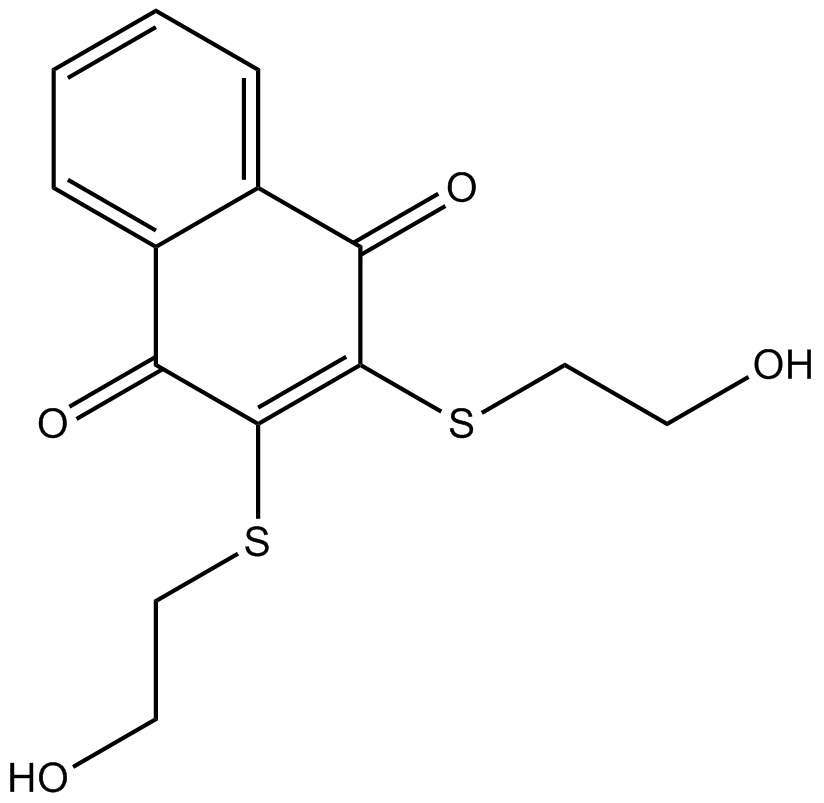
-
GC14103
NSC228155
NSC228155 est un activateur de l'EGFR, se lie à la région extracellulaire de l'EGFR et améliore la phosphorylation de la tyrosine de l'EGFR. NSC228155 est également un puissant inhibiteur de l'interaction KIX-KID, inhibe le domaine inductible par la kinase (KID) de CREB et le domaine interagissant avec le KID (KIX) de CBP, avec une IC50 de 0,36 μM.